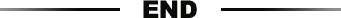广州市黄埔区学大道揽月路广州企业孵化器B座402
电话:020-85625352
手机:18102256923、18102253682
Email:servers@gzscbio.com
Fax:020-85625352
QQ:386244141
HMGNs对REX1表达和染色质结合特异性的表观遗传调控
日期:2020-03-04 标签:HMGNs,REX1

摘要
HMGN蛋白位于染色质调节位点,并调节细胞类型的特异性转录谱。然而,这些核小体结合蛋白影响基因表达的分子机制尚不完全清楚。作者在描述了HMGNs通过将转录因子NANOG,OCT4和SOX2募集到位于第5位的ESC特异性超级增强子,来调节Rex1的表达,Rex1是小鼠胚胎干细胞(ESC)中转录度最高的基因之一。在Rex1的区域,HMGN有助于建立活性染色质和增强子启动子相互作用的表观遗传模型,如下图所示:HMGNs的丢失会改变局部表观遗传谱,增加组蛋白H1的占有率,降低转录因子的结合并降低增强子启动子的互作,从而下调Rex1的表达。ChIP-seq分析显示,在启动子和增强子上,HMGNs和锌指蛋白REX1的色泽很高。 HMGNs的丢失会优先降低REX1与这些染色质调节位点的特异性结合。因此,HMGNs影响REX1的表达和染色质结合特异性。我们建议HMGNs通过调节转录因子对色母蛋白的结合特异性来影响细胞类型的特定基因表达。
![]()
图1.HMGN与染色质互作和对Rex1表达影响的模型。染色质环是由位于基因座上游和下游区域两侧的CTCF分子互作形成的,Rex1基因座位于染色质环内。在WT细胞(图的左侧)中,位于5位超级增强子上的NANOG,OCT4和SOX2与Rex1基因的结合促进了与近端调控元件的相互作用,从而促进了Rex1表达。HMGN的丢失(右侧)不会影响CTCF结合,但会增加H1的占有率,解除转录因子与超级增强子的结合,并降低DNase I敏感性和标记活性染色质的组蛋白修饰水平。这些表观遗传学的变化可以防止Rex1增强子和Rex1近端调节位点之间的接触,从而下调Rex1的表达。
01
HMGN蛋白调节小鼠胚胎干(ES)细胞中Rex1基因的表达
作者进行LIF / 2i(可使悬浮生长的小鼠ESC保持其多能性)去除后的第0、2、4、6天基因表达分析,显示与WT相比HMGN DKO中176、239、447和592个基因的表达下降而只有13个基因始终被下调(图2A)。表明,在每个分化阶段,HMGNs调节一组独特基因的表达。在DKO细胞中始终被下调的13个基因中,作者研究了Rex1(Zfp-42,一种已知会影响多能性的锌指蛋白)。Q-pcr显示Rex1在小鼠胚胎干细胞中高表达,在LIF / 2i去除后的第2天,ESC中Rex1表达被显着下调(图2B),在HMGNs DKO ESC中,Rex1的表达比野生型ESC中的表达低3倍以上,并且在EB分化的所有6天中,DKO细胞中Rex1的转录仍然较低(图2B和C)。相反,HMGNs的丢失对管家基因如Actb的表达几乎没有影响(图2D)。WB也证实DKO中的REX1蛋白水平比WT细胞低50%以上(图2E)。说明Rex1转录本的下调归因于HMGN的丢失。
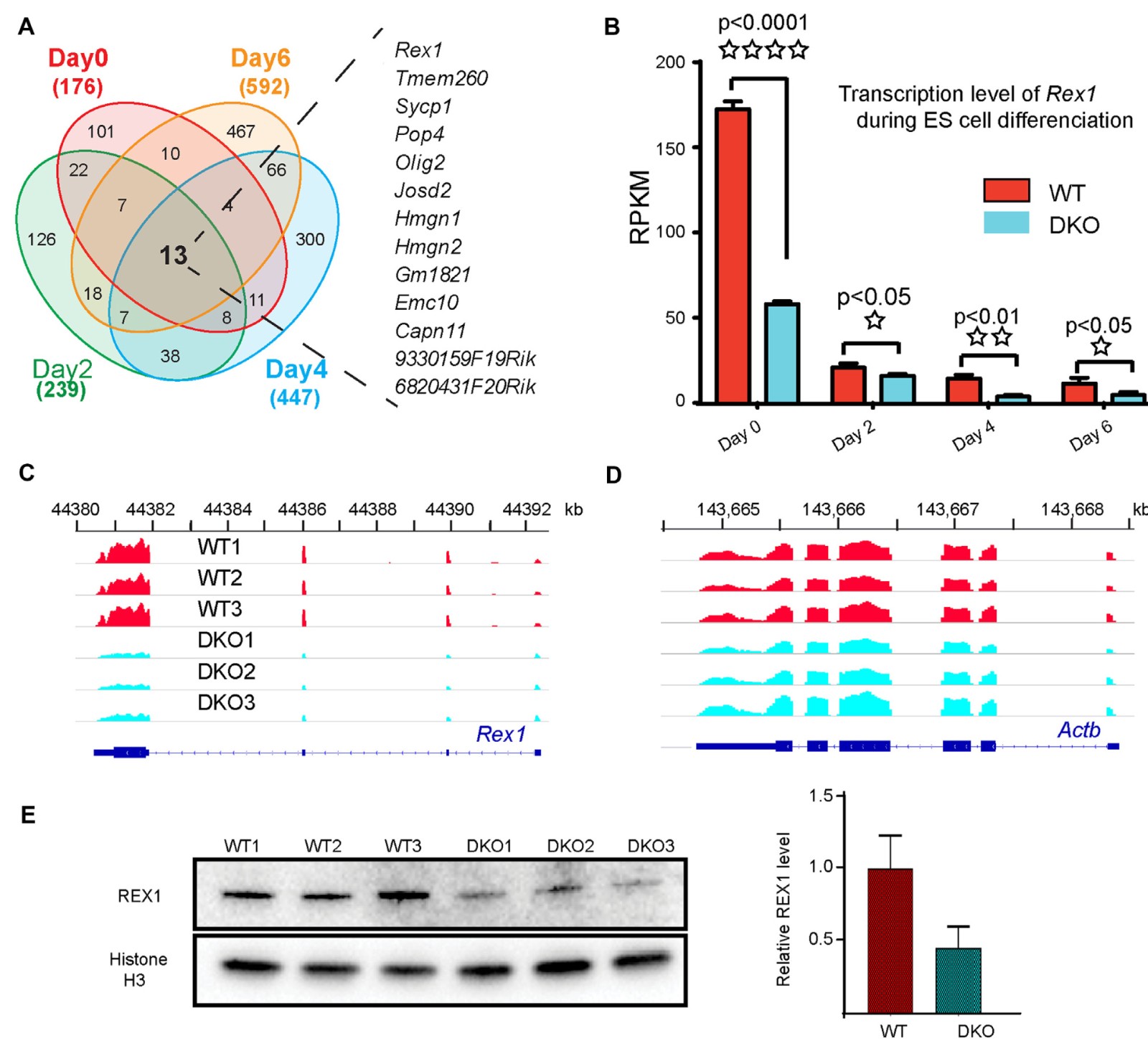
图2. HMGN蛋白影响Rex1表达。
(A)维恩图显示了在LIF / 2i去除后的不同天,与WT胚胎干细胞相比,DKO中下调基因之间的重叠。图的外围显示了在每个EB分化阶段DKO中表达下调的基因总数(1.3倍; P <0.05)。图的右边列出的13个基因在整个分化过程中均被下调。
(B)在LIF / 21退出后指定天数后通过mRNA-seq分析WT和DKO ESC中Rex1的转录水平。(C)3个生物学复制的IGV数据,显示ESC中Rex1的mRNA水平。
(D)IGV显示Actb的mRNA水平作为对照。
(E)WB检测ESC中REX1的表达。
02
HMGN蛋白的丢失会改变Rex1调控位点的染色质结构
为了了解HMGNs调节Rex1表达的机制,作者首先检测了HMGNs的丢失是否改变该基因的调控区域的染色质结构。通过HMGN1和HMGN2的ChIP-seq分析显示在Rex1基因的5个区域和两个富含H3K27ac和H3K4me1的上游区域有较高的HMGN占用率。位于距Rex1基因14 kb处的远端(图3中的棕星)显示出较高的DNaseI相对敏感性,这对应已知的ESC特异性增强子。HMGN的缺失导致DNaseI敏感性以及H3K27ac和H3K4me1水平显着降低(图2中的箭头),会导致基因H3K27ac和H3K4me1(箭头)以及H3K4me3,H3K9Ac和H2BK5ac在该基因的最接近5区的水平降低(图3中的虚线框),而这些转录活性染色质标记的下调不如在远端超级增强子区域看到的明显。总之,我们对WT和DKO ESC的ChIP-seq分析表明,HMGN蛋白的缺失降低了Rex1调控位点的活性染色质标记和DNase I超敏性水平。
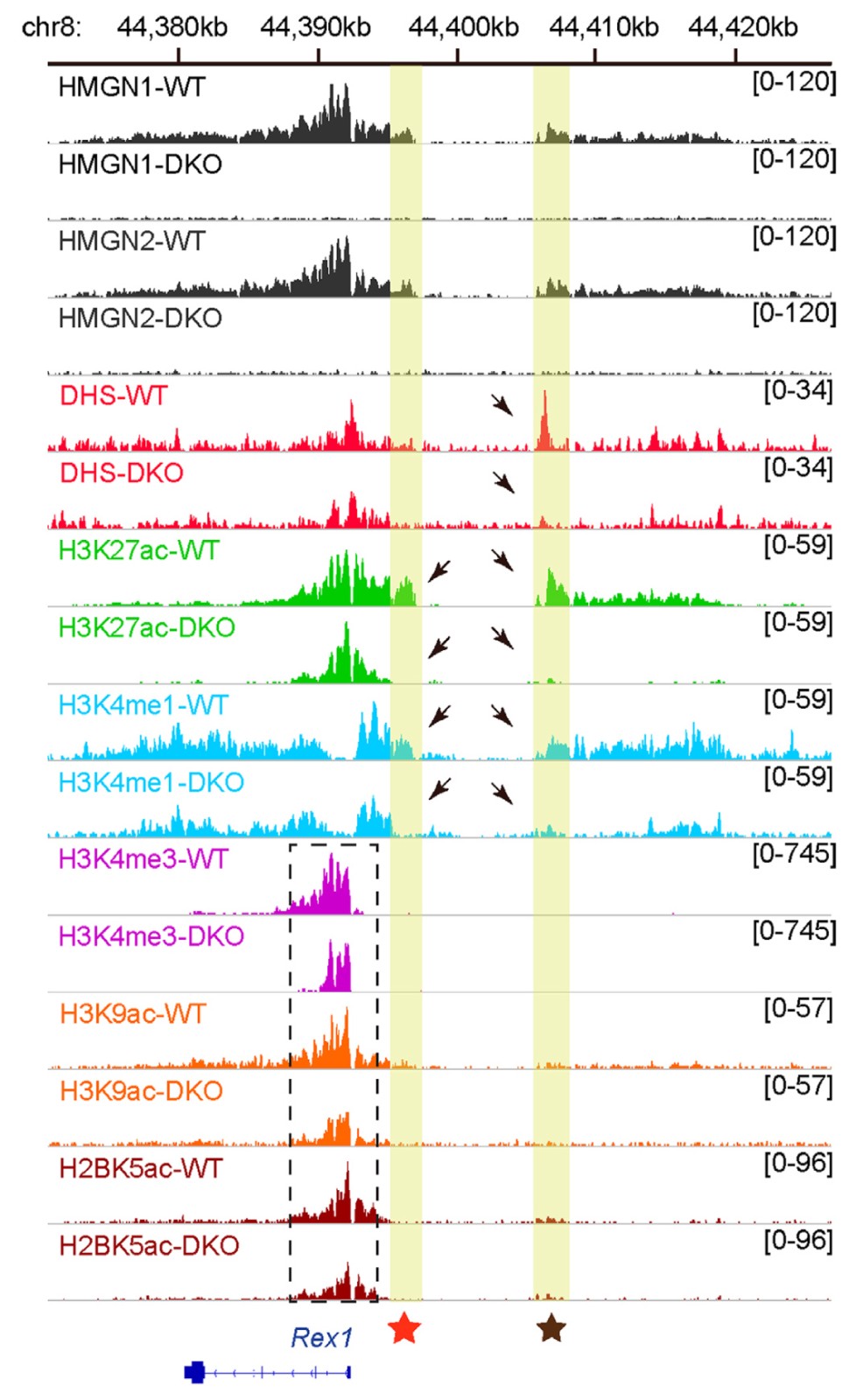
图3. HMGN的缺失改变了Rex1基因座的染色质结构。显示的是IGV,描述了HMGN1和HMGN2的占有率,DNase I超敏性(DHS)以及从WT和DKO小鼠中分离出的ESC的Rex1基因座上所示的组蛋白修饰水平。 黄色列突出显示了近端(红星)和远端(棕星)增强子区域的位置。箭头指向DKO细胞中DNase1,H2K27ac和H3K4me1的丢失,Rex1基因近端5区的表观遗传变化在虚线框内可见。
03
HMGN蛋白的丢失减少了OCT4,NANOG和SOX2转录因子与Rex1超级增强子的结合,增强了组蛋白H1与Rex1超增强子的结合
已知ESC中Rex1的表达受转录因子OCT4,NANOG和SOX2调控。mRNA序列分析表明HMGN蛋白的丢失不会影响Oct4,Nanog或Sox2的转录水平(图4A),因此HMGN可能是通过调节转录因子的结合来调节Rex1表达。ChIP-seq分析显示,位于Rex1基因5处的超级增强子区域(图4B,棕星)处的所有三个转录因子均具有较高的占有率,而在近端增强子区域则没有(图4B红星),HMGN的丧失完全消除了OCT4,NANOG的结合,并降低了SOX2与Rex1超级增强剂的结合(图4A,箭头)。H1促进并稳定染色质的缩合,可能降低染色质调节位点的活性。为了确定HMGNs是否影响组蛋白H1染色质的占有率,用H1抗体ChIP-qPCR (图4C)。在WT中,与Rex1超增强子重叠的所有三个区域中H1的占有率明显低于与相邻非调节区域重叠的五个区域中的每个区域中的占有率(比较图4D中的蓝色条)。HMGN的丢失会导致Rex1超级增强器中三个位置的每个位置的H1占用显着上调(图4D)。 WT细胞中非调节位点H1的平均占有率与DKO细胞中的相同,但在超增强位点,HMGN的丢失导致H1占有率增加了50%(图4E)。这解释了该调控位点转录因子占有率降低和活性染色质的表观遗传标记的丧失。
图4. HMGN-H1互作调节转录因子与Rex1超级增强子的结合。
(A)mRNA-seq,HMGN的丢失不影响Oct4,Nanog或Sox2的表达。
(B)IGV显示DKO ESC中Rex1超级增强子的OCT4,NANOG和SOX2结合丢失。红色和棕色的星星分别表示近端增强子和超级增强子区域的位置。箭头指向转录因子的位置,在底部显示的这些调控区域中选择组蛋白标记。
(C)选择用于组蛋白H1 ChIP-qPCR实验的基因组区域。
(D)q-pcr检测C中所示Rex 1的五个非调节区域和三个超级增强器区域中WT和DKO ESC中的H1相对占有率。(E)DKO中Rex1超级增强子的H1占有率升高,但相邻的非调节染色质区域中不变。
04
HMGNs促进Rex1启动子和增强子之间的相互作用
先前的ChIA PET结果揭示了Rex1远端超级增强子与Rex1启动子之间存在显着互作(图5A),从而增加了连接这些调控位点的染色质环控制Rex1表达水平的可能性。为了测试这种可能性,作者进行3C实验分析,并比较了WT和DKO ES细胞中Rex1启动子-增强子的互作。3C文库的PCR扩增显示,靶向5个上游Rex1调控区域产生了一条清晰的条带,表明调控因子之间的互作频率很高,相反,相同的引物组不能扩增从DKO细胞生成的3C文库(图5C)。 Sanger测序证实,源自WT文库的PCR产物由与Rex1增强子和启动子区域相邻的DNA序列组成(图5D)。总之,ChIA-PET和3C实验均表明,在WT细胞的染色质中,位于该基因5个近端区域的Rex1调控位点与位于14kb转录起始位点上游以上的远端超增强子互作,而在缺乏HMGN的DKO细胞中则没有。
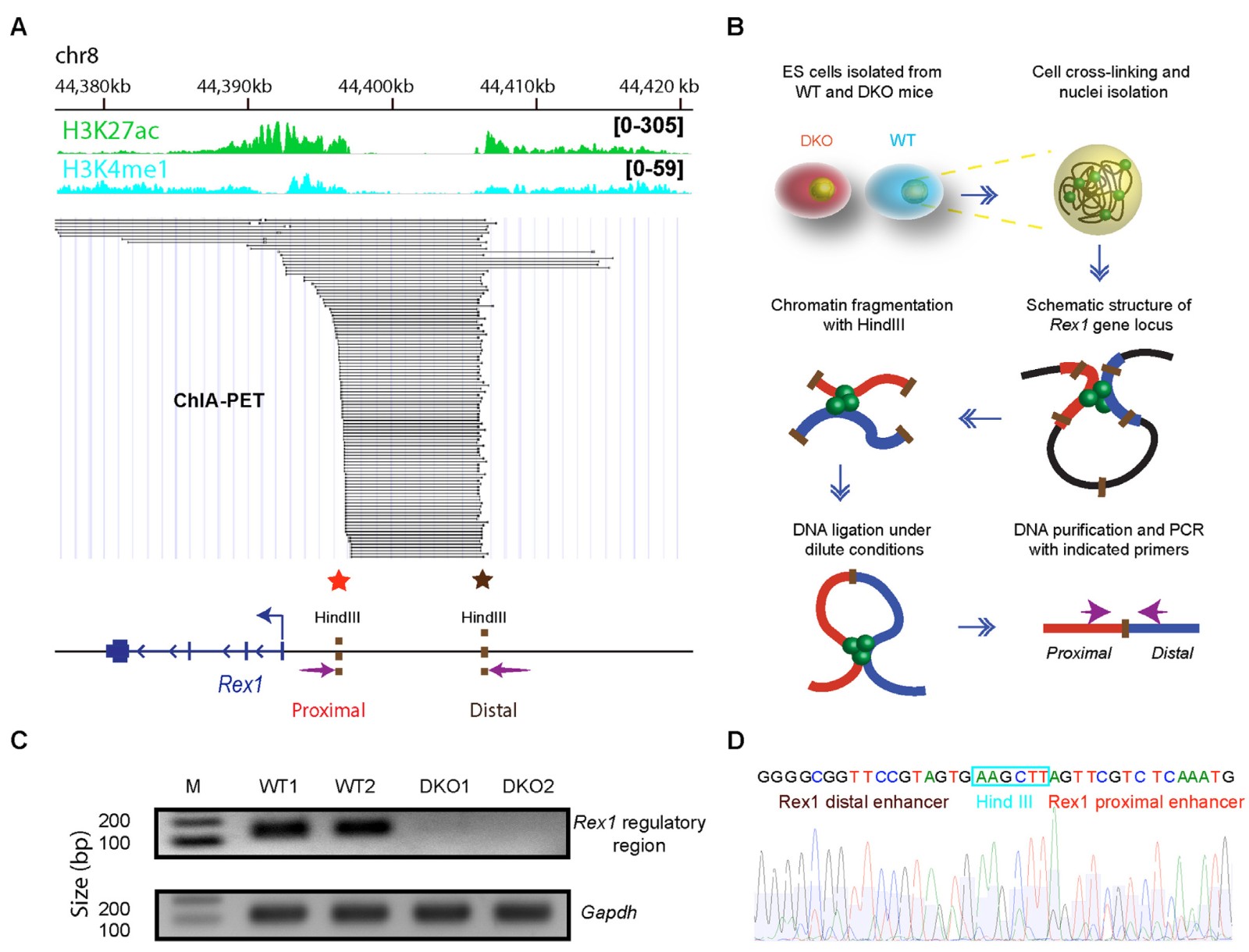
图5. HMGN的缺失破坏了Rex1 5调控区的染色质互作。
(A)Rex1基因座上染色质调节区之间相互作用的ChIA-PET分析。顶部显示管制区域和所选的组蛋白标记。图中的每条线表示两个基因组基因座之间检测到的相互作用。
(B)用于检测Rex1的5个调控区中的染色质互作的3C实验示意图。
(C)使用位于Rex1的5个调节子区域的引物对3C文库进行PCR扩增得到的DNA片段。
(D)Sanger测序证实PCR产物对应于位于Rex1的5个区域中调节位点附近的两个区域的连接。