广州市黄埔区学大道揽月路广州企业孵化器B座402
电话:020-85625352
手机:18102256923、18102253682
Email:servers@gzscbio.com
Fax:020-85625352
QQ:386244141
如何通过3D基因组研究结构变异
日期:2018-11-15 标签:结构变异,位置效应,3D基因组测序技术,Hi-C
最近研究表明,结构变异(structural variation,SV)不仅可以影响基因剂量,而且是基因调控的基本机制。SV可以改变调控元件的拷贝数,或者通过破坏染色体的高级结构如TAD来影响基因组的3D结构。由于这些位置效应,SV可以影响离SV断点很远的基因表达,从而导致疾病的发生。SV对3D基因组和基因表达调控的影响,已经被认为是导致疾病发生的潜在原因。
结构变异(SV)包括删除(deletions),重复(duplications),倒位(inversions),插入(insertions)和易位(translocations)。高通量测序技术大大加快了发现和表征这些SVs,然而,在医学上解释SVs和疾病表型结果,仍然不尽人意。SVs可以在不改变编码基因序列情况下导致疾病的发生,这暗示着SV可能作为一个调控原件发挥作用,即所谓的位置效应。事实上,基因组98%的区域都是非编码区域,因此大部分非编码区域可能以某种方式参与基因的调控,但是具体有多少区域参与调控仍然不清楚。因而SV通过影响顺式调控元件(如启动子和增强子)的位置和/或功能,参与基因的调控,成为了非编码区域调控的热点。最近随着3D基因组测序技术发展,位置效应作为调控的热点的证据越来越明显。因而小编坚信接下来用Hi-C鉴定结构变异,揭示基因调控原理,阐明性状产生的根本原因必然是动植物基因组结构变异研究的热点。下面具体介绍一下Hi-C做结构变异的原理。
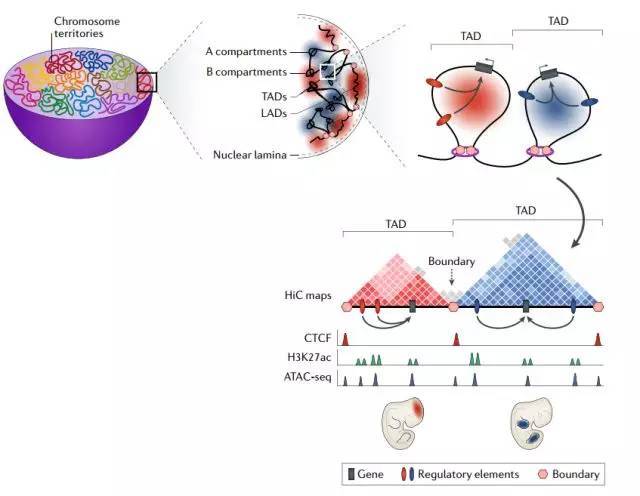
在细胞核级别,染色体占据特定的领域(下图)。在染色体水平上,Hi-C技术已经揭示了染色质可以分为开放和闭合的空间,称为A/B隔室。A与可接近的,转录活性的常染色质紧密相关,B与不可接近的,转录沉默异染色质相关(见图)。随着测序深度和分辨率的增加,隔室被细分为为拓扑结构域(TADs)。在Hi-C实验中TADs鉴定主要依据在边界地区交互作用减少(见图)。在TADs内部,调控元件与调控的基因相互作用,激活基因的转录。组蛋白H3K27ac和ATAC-seq可用于鉴定活性调控元件(见图)。TAD边界通过粘连蛋白复合物来稳定,并且通常富含CTCF和转座元件或看家基因;边界对TAD行使正常功能是至关重要的。
在更高的分辨率下,Hi-C数据集已经揭示了额外层级结构的存在,例如sub-TADs,它们是较小的空间域(大约100kb),显示出更加动态的性质和组织特异性。此外,Loop(环)位于TAD和sub-TAD中,可以在HiC热图中直观识别。一些环与增强子-启动子相互作用有关,也显示出动态性和组织特异性。
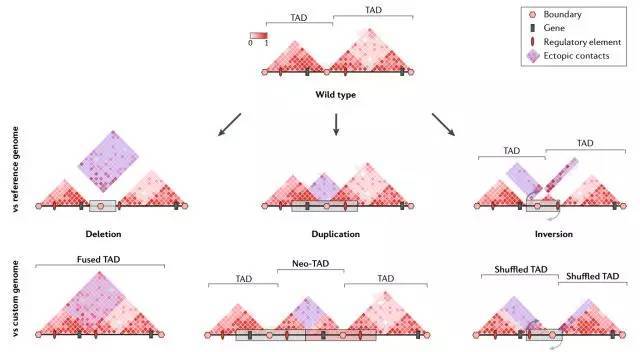
结构变异可以诱发染色质组织结构的巨大变化,从而产生特定的特征,通过交互热图可以发现明显的特征。示意图显示不同类型结构变异导致Hi-C热图变化。通过与野生型参考Hi-C热图进行比较(上图),由空间邻近变化引起的异位相互作用变得明显(用蓝色表示);基于参考基因组的Hi-C热图提供变异的特定信息(即删除,重复,倒位或易位;用灰色矩形标记)以及单核苷酸分辨率的断点位置。此外,通过将Hi-C数据映射到含有SV的基因组(下图),可观察到潜在的病理机制,如TAD融合(缺失),neo-TAD形成(复制)或TAD重组(倒位)。
3D基因组中SV与疾病
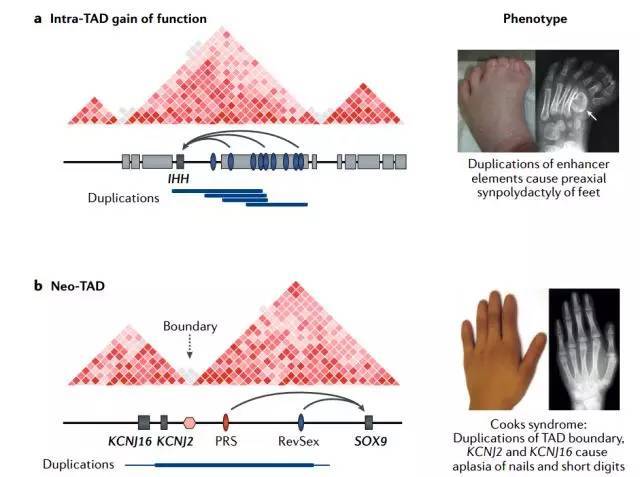
a、在TAD内IHH基因座的增强子的复制,导致该基因表达发生组织特异性失调并与脚的多趾症相关。
b、SOX9位点的边界复制,导致的TAD形成,并与烹调综合征、短指有关。
c、删除LMNB1基因座附近的TAD边界导致另一个TAD内部增强子调控LMNB1基因,进而导致成人发作性脱髓鞘性脑白质营养不良。
d、翻转EPHA4基因座增强子簇,导致增强子错误调控WNT6,导致拇指和食指并指畸形。
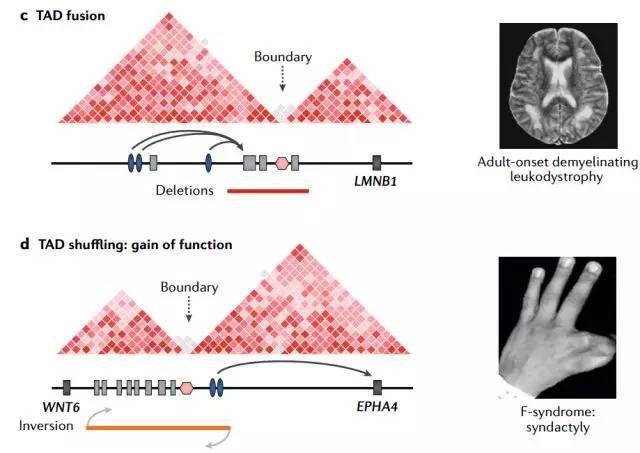
e、平衡易位。MEF2C易位,将会失去调节元件,导致脑部异常和发育迟缓
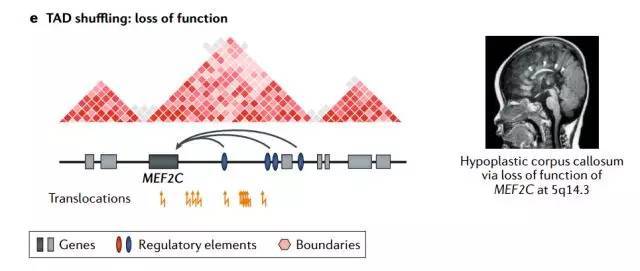
TAD内部结构变异(SV)通过改变增强子的剂量效应,可能造成功能的丢失或增加。
a、野生型示意图中基因A在发育中的大脑中表达和基因B在肢体中表达。两个基因分别由他们自己的组织特异性的顺式调控元件调控(分别为红色和蓝色,位于不同的TAD中)
b、TAD内倒位不破坏编码基因或TAD边界,对基因的远程调控无重大影响,目前没有病例报告。
c、TAD内缺失增强子元件,可导致基因B在肢体不表达。
d、TAD内部复制。调控元件复制,基因B组织特异性的肢体过度或者错误表达(蓝色),进而导致疾病。
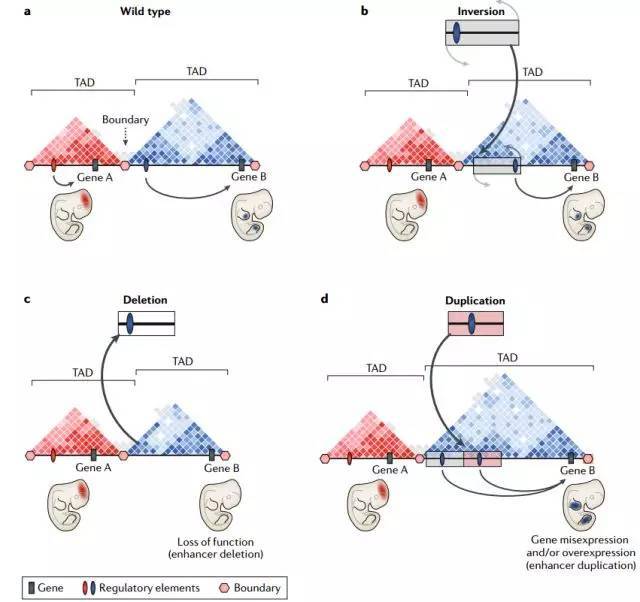
1)删除和重复类型的SV。
TAD边界结构的变化会扰乱染色质结构域和形成新的调控单元。
a、野生型示意图中基因A在发育中的大脑中表达和基因B在肢体中表达。两个基因分别由他们自己的组织特异性的顺式调控元件调控(分别为红色和蓝色,位于不同的TAD中)。
b、边界元间的TAD删除能引起TAD的融合;基因B的增强子会调控基因A,导致基因A错误在肢体中异位表达和疾病产生。
c、TAD边界发生复制,形成一个新的TAD。在新的TAD中点,发生复制的基因A在发生复制的B基因的增强子的控制下,在四肢中异位表达,并导致疾病发生。
d、不包括基因A的TAD边界复制,也形成了新的TAD,但尚未发生转录变化。
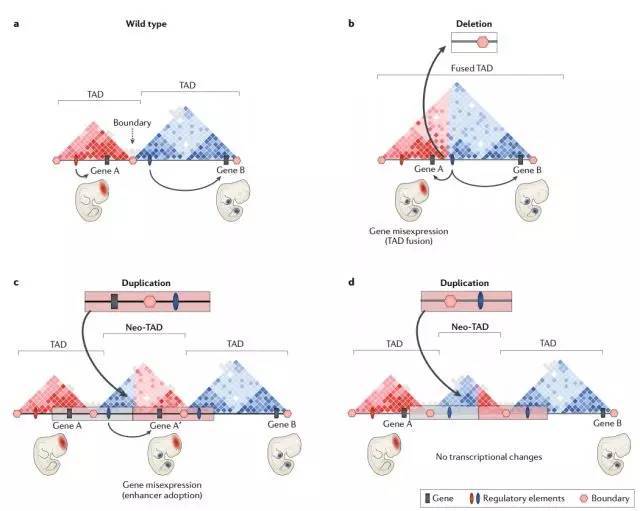
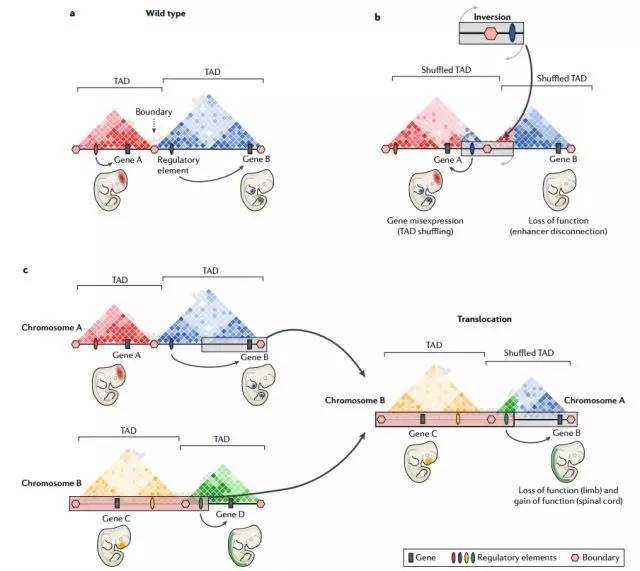
2)倒位和易位类型的SV
b、基因B的增强子倒位,导致A基因在肢体中异位表达。同时,基因B缺乏增强子,导致基因B不表达,进而导致肢体功能的调节性丧失。
c、平衡易位。位于染色体A的基因B与位于染色体B的基因D发生易位,重组后形成了c图中右半部分。由于基因B丢掉增强子,获得基因D的增强子,因而导致B基因在肢体中的表达缺失,在脊髓的异位表达。易位导致的结果取决于断点和侧翼基因。易位也可能是不平衡的。
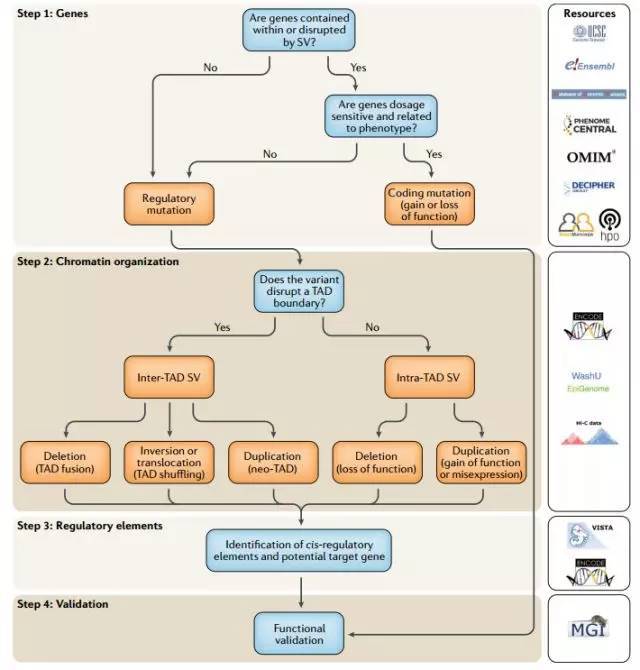
从3D基因组角度研究SV的策略
首先识别有拷贝数变异或SV变异的基因(步骤1)。如果重排是平衡的,识别可能被破坏的基因。检查基因是否剂量敏感和/或表型相关。如果基因没有位于重排区域或者不是剂量敏感,从Hi-C交互热图判断SV位于TAD内部还是TAD边界。根据所示原理对SV进行分类(步骤2)。识别潜在的可以驱动一个疾病基因异位表达增强子(步骤3)。如果可能的话,计算异位增强子–启动子相互作用与基因的表达(肿瘤)或表型(先天性疾病)关系(步骤4)。解释结果,如有必要,进行功能验证。