
广州市黄埔区学大道揽月路广州企业孵化器B座402
电话:020-85625352
手机:18102256923、18102253682
Email:servers@gzscbio.com
Fax:020-85625352
QQ:386244141
项目名称:差异Peak计算
所属分类:生物信息学分析
联系电话:020-85625352
QQ:386244141
Email:servers@gzscbio.com
技术服务描述
1. 差异Peak计算
1.1. 背景简介
为了理解细胞中更为复杂的生物过程,许多研究已在通过比较ChIP-seq的差异获得的不同数据。
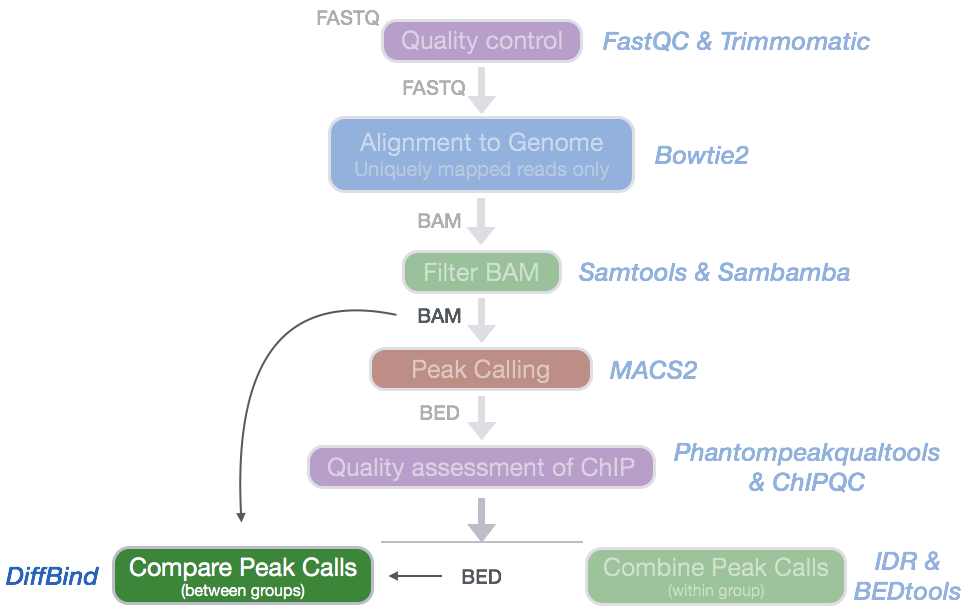
越来越多的ChIP-seq实验正在研究多种实验条件(例如各种治疗条件,几个不同的时间点和不同的治疗剂量水平)下的转录因子结合。由于差异富集在生物学和医学研究中已变得具有实际重要性,因此已有更多工具可用于此类分析。
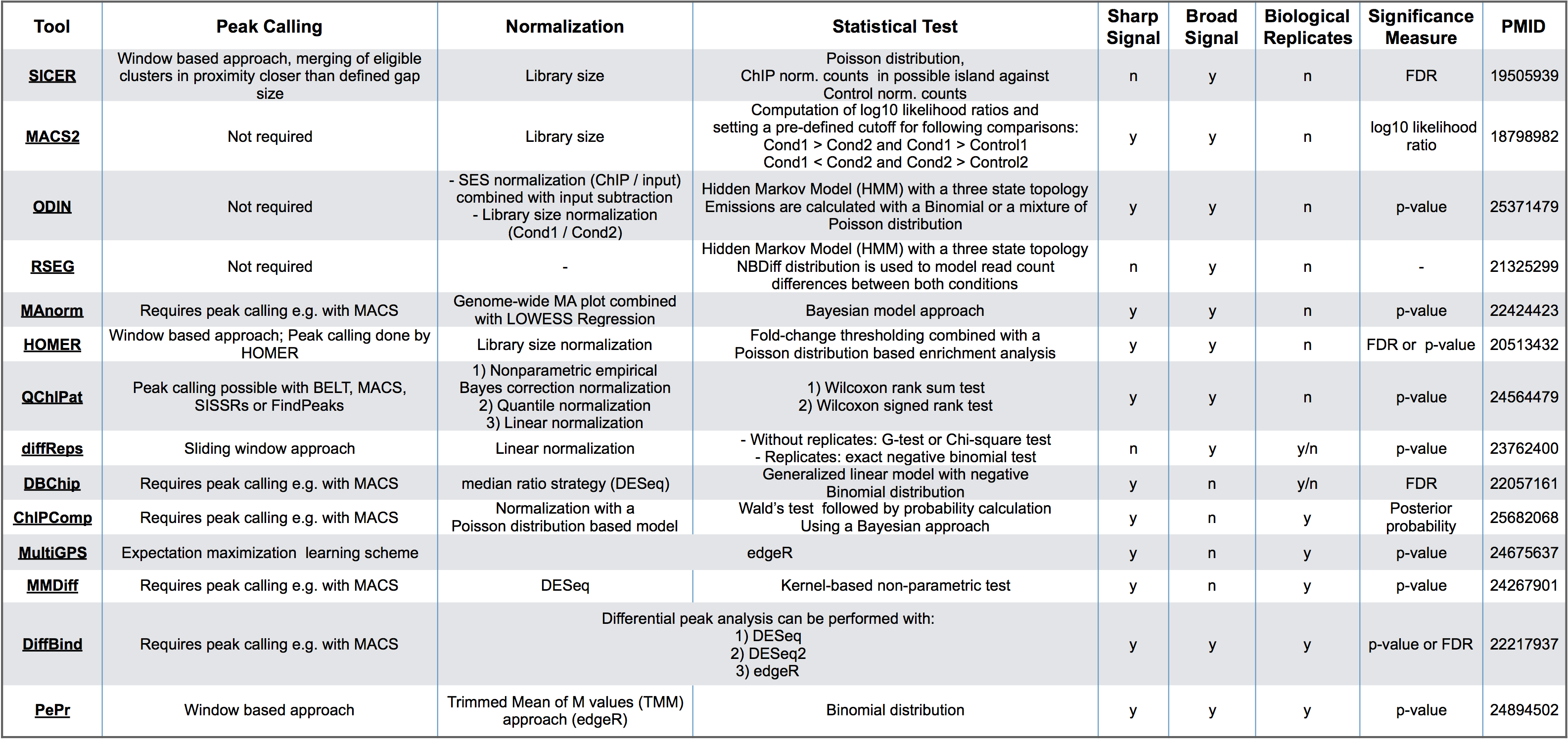
每种工具的工作方式互不相同,但总体目标相同。您选择的一个将归结为您正在使用的数据集以及您的生物学问题。要考虑的一些事情: - 用户需要什么输入?一些工具需要通过外部峰调用算法对富集区域进行初步检测,而其他工具则实现了自己的检测方法。 - 该工具是否有助于在每个样本组中使用重复样本? - 用于信号分配的基础统计模型是什么?它是基于泊松分布还是基于更灵活的负二项式分布。 - 已经针对特定的ChIP-seq数据(信号类型)专门设计了一些工具,例如组蛋白修饰或转录因子(TF)结合。
对于有两个重复的样本,最好使用DiffBind之类的工具来利用这些重复项。
1.1. DiffBind
DiffBind是一种R Bioconductor软件包,用于识别两个或多个样品组之间差异富集的位点。 它主要与峰调用集(“峰集”)配合使用,这是代表每个样品的候选蛋白质结合位点的基因组间隔集。它包括支持峰集处理的功能,包括在整个数据集中重叠和合并峰集,对峰集中重叠间隔中的测序读数进行计数,并根据结合亲和力的证据在统计学上显着鉴定差异结合位点(通过读数差异来衡量)密度)。
1.2. DiffBind的基本计算步骤介绍
1.2.1. 阅读峰集
第一步是读取一组峰集和关联的元数据。这是使用样本表完成的。读入峰集后,合并功能会找到所有重叠的峰,并得出覆盖所有提供的峰的单组独特的基因组间隔(实验的共识峰集)。如果一个区域出现在两个以上的样本中,则认为该区域为共识集。该共有集代表将在进一步分析中使用的候选结合位点的总体集。
简言之:寻找两组对比样本中的共有Peak区域。
1.2.2. 亲和力结合矩阵
下一步是获取比对文件,并为共识集中的每个峰/区域计算计数信息。在此步骤中,对于每个共有区域,DiffBind会获取ChIP样本和输入样本中对齐的读取数,以计算每个潜在结合位点上每个样本的标准化读取数。共识峰集中的峰可以基于计算其峰顶(最大读取重叠点)进行重新居中和修整,以提供更标准的峰间隔。
1.2.3. 探索性数据分析
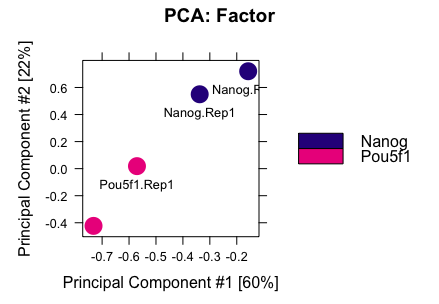
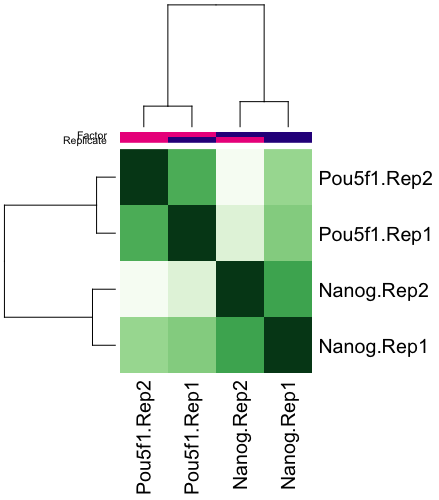
1.2.3.1. Peak共有位点相关性分析
为了了解样本之间的聚类程度,我们使用所有共有位点绘制PCA图,相关性热图
1.2.3.2. 差异分析
DiffBind的核心功能是差异结合亲和力分析,该功能可识别出在样品组之间统计学上显着差异结合的结合位点。默认情况下,使用DESeq2执行核心分析例程,并且还可以选择使用edgeR。每个工具将为每个候选结合位点分配一个p值和FDR,以表明它们被差分结合的置信度。
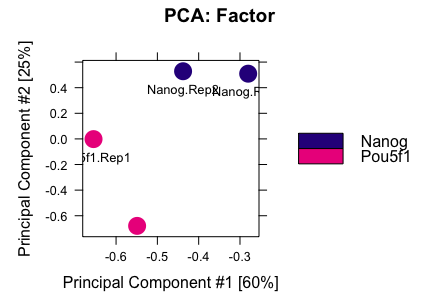
1.2.3.2.1. 差异Peak的相关性分析
为了了解样本之间的差异Peak计算结果的重复性,我们使用所有差异分析结果绘制PCA图,相关性热图
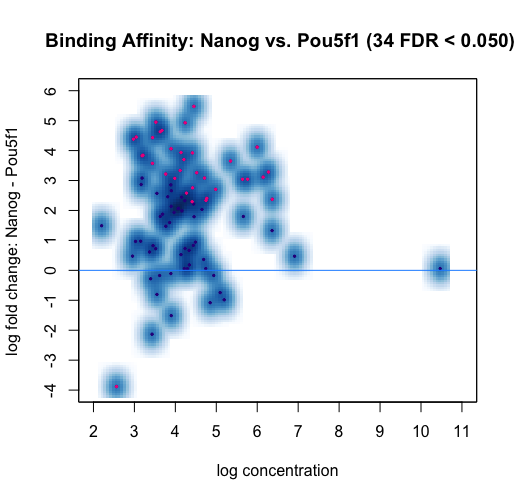
MA图
差异Peak一种可视化方法。
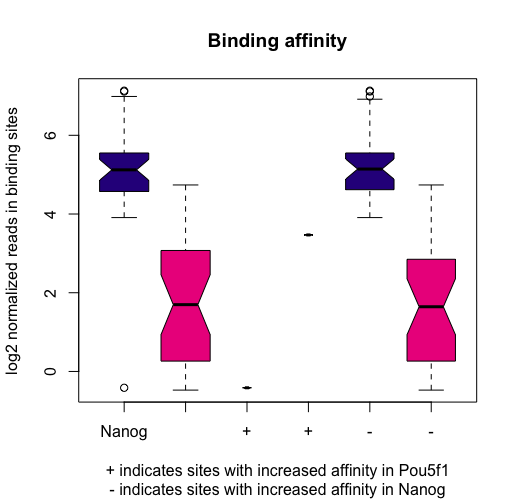
箱型图
显示在样品中表现出增加的亲和力的差异结合位点中的reads分布。
结果
表头说明 【 之前给的分析报告里面有,待抄过来 】
Demo
1.1.chip/DiffBind/Demo_analysis/
| 文件 | 说明 |
|---|---|
| ZGW_H3K27ac/1cor_readCont_ZGW_H3K27ac.png | 读取的reads计数的相关性分析(热图) |
| ZGW_H3K27ac/1pca_readCont_ZGW_H3K27ac.png | 读取的reads计数的相关性分析(PCA) |
| ZGW_H3K27ac/2cor_peakScore_ZGW_H3K27ac.png | Peak共有位点相关性分析(热图) |
| ZGW_H3K27ac/2pca_peakScore_ZGW_H3K27ac.png | Peak共有位点相关性分析(PCA) |
| ZGW_H3K27ac/ZGW_H3K27ac_treat-vs-con_1cor.png | 差异Peak可视化,相关性分析(热图) |
| ZGW_H3K27ac/ZGW_H3K27ac_treat-vs-con_2pca.png | 差异Peak可视化,相关性分析(热图) |
| ZGW_H3K27ac/ZGW_H3K27ac_treat-vs-con_3ma.png | 差异Peak可视化,MA图 |
| ZGW_H3K27ac/ZGW_H3K27ac_treat-vs-con_4vol.png | 差异Peak可视化,火山图 |
| ZGW_H3K27ac/ZGW_H3K27ac_treat-vs-con_5box.png | 差异Peak可视化,关于reads分布的箱型图 |
| ZGW_H3K27ac/ZGW_H3K27ac_treat-vs-con_6heatmap.png | 差异Peak可视化,表达热图 |
