
广州市黄埔区学大道揽月路广州企业孵化器B座402
电话:020-85625352
手机:18102256923、18102253682
Email:servers@gzscbio.com
Fax:020-85625352
QQ:386244141
项目名称:染色质免疫共沉淀测序 ChIP-Seq 结题报告
所属分类:生物信息学分析-报告解读
联系电话:020-85625352
QQ:386244141
Email:servers@gzscbio.com
技术服务描述
染色质免疫共沉淀测序 ChIP-Seq 结题报告
生信部
2020年08月25日
项目信息
合同编号:XXX-2020-01-01-01
客户姓名:XXX
客户单位:X X X X X X
1. 工作流程
染色体免疫共沉淀(ChIP)是一种用于研究蛋白质与 DNA 的体内相互作用的经典实验技术。采用特异性抗体将目的蛋白进行免疫沉淀,由此可以把目的蛋白所结合的基因组 DNA 片段也富集下来。通过与高通量测序技术的结合,对 ChIP 后的DNA 产物进行测序分析, 从全基因组范围内寻找目的蛋白的 DNA 结合位点,以高效率的测序手段得到高通量的数据结果。
1.1. ChIP 免疫沉淀实验流程
目前主要有两种不同的ChIP 实验方法,大致流程如下(以细胞样品的处理过程为例):
Cross-liking Chromatin Immunoprecipitation (X-ChIP)
准备足量的新鲜细胞,每个IP约4x106个细胞,用新鲜的1%的甲醛处理细胞,进行细胞交联。
125mM的甘氨酸终止交联,收集细胞。
超声或酶解打断染色质,将基因组 DNA 打断至 100-500bp。
将抗体(一般为1~5ug)与染色质片段4℃孵育过夜。
加入proteinA/G beads进行4℃孵育4-6小时。
Proteinase K 解交连。
酚氯仿或DNA提取试剂盒提取DNA
QPCR 检测或建库测序
1.2. ChIP Sequencing 文库构建流程
用qubit 对ChIP片段进行定量检测
补齐片段末端,并在3’末端加A尾
添加Adapter
0.8X AMPure beads去掉多余的Adapter
文库PCR扩增
1XAMPure beads 去掉多余的primer
qPCR测定文库浓度
Agilent 2100测定文库片段大小
1.3. 生物信息分析流程
将测序结果与参考基因组比对,比对上唯一位置的序列用于后续标准信息分析及个性化分析。信息分析流程如下:

2. 数据结果及生物信息分析
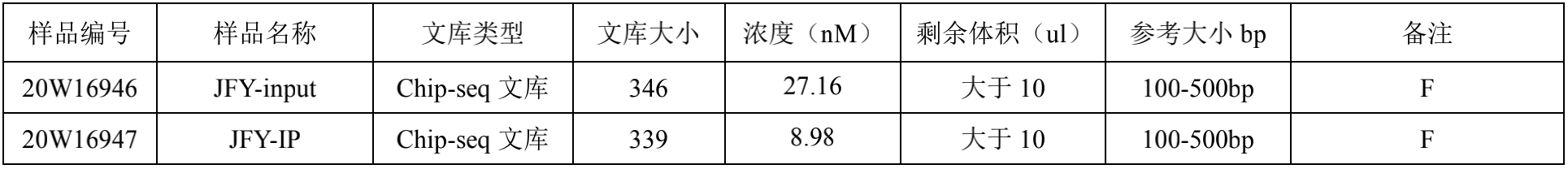
2.1. ChIP Sequencing 文库质检结果
文库片段质检,ChIP文库的染色质片段在100-500bp之间,建库加入约140bp的接头后,片段应该分布在250-700bp之间为最好。
Fragment Analyzer (FA)毛细管电泳检测:

检测结果汇总:(以下结果中文库大小为 FA 判定结果)
2.2. 测序数据质量控制
对原始测序数据及去除接头后的可用数据进行质量评估。
具体的qc报告见:
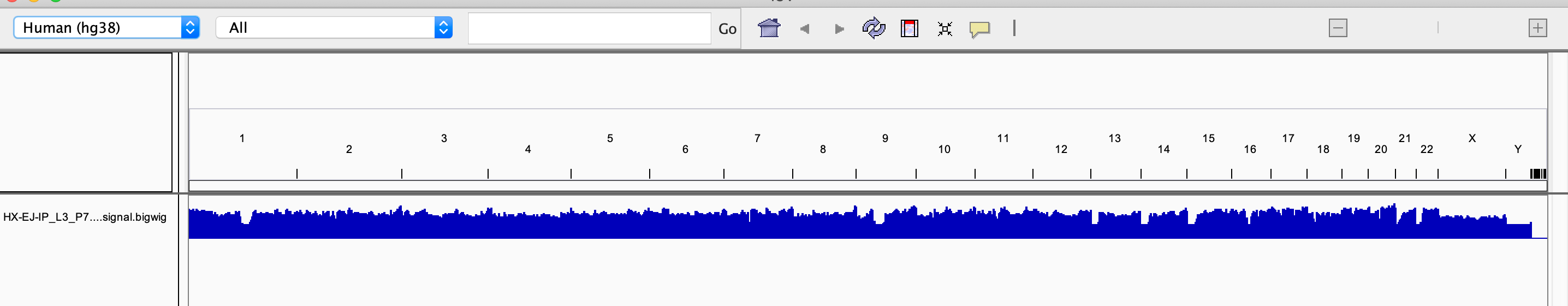
2.3. Reads 在全基因组的可视化分布
使用 IGV 软件对 Reads 进行可视化查看,可以查看全基因组任何感兴趣位置的 reads 富集情况,示例如下:
![]()
IGV 的安装使用参考:http://software.broadinstitute.org/software/igv/
可视化操作步骤依次是:
在软件的 Genome 选项,基因参考序列 hg38 ;
在软件的 File 选项,上传 要查看染色体 bigwig 文件;
以上文件上传后可查看该染色体任意位置的基因信息及 reads 富集情况。
结果文件 :
2.4. 全基因组 Reads 富集峰 Peak 鉴定
采用常用 reads 富集峰鉴定软件 MACS 在全基因范围进行 peak 扫描,得到 Peak 在基因组上的位置信息、peak 富集信息等。
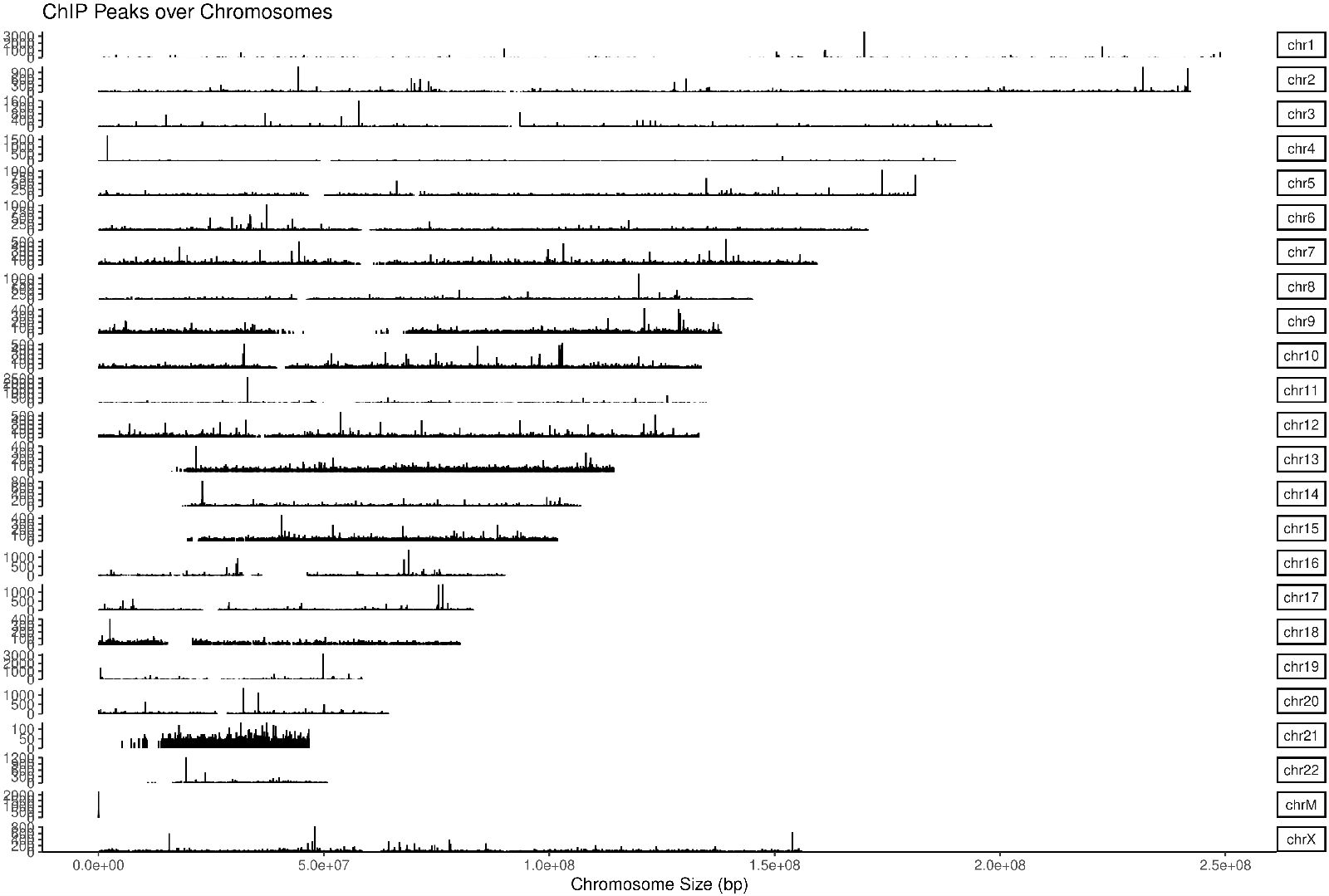
图1 全基因组 Reads 富集峰
结果文件:
Results/JFY.peakanno.csv
Results/2.4.peak_scan/JFY.covplot.pdf
2.5. Reads 在 TSS 近端富集强度分析
TSS 转录起始位点近端(0-3kb)与特定的基因转录调控功能有关,统计 reads 在TSS 近端的分布情况。
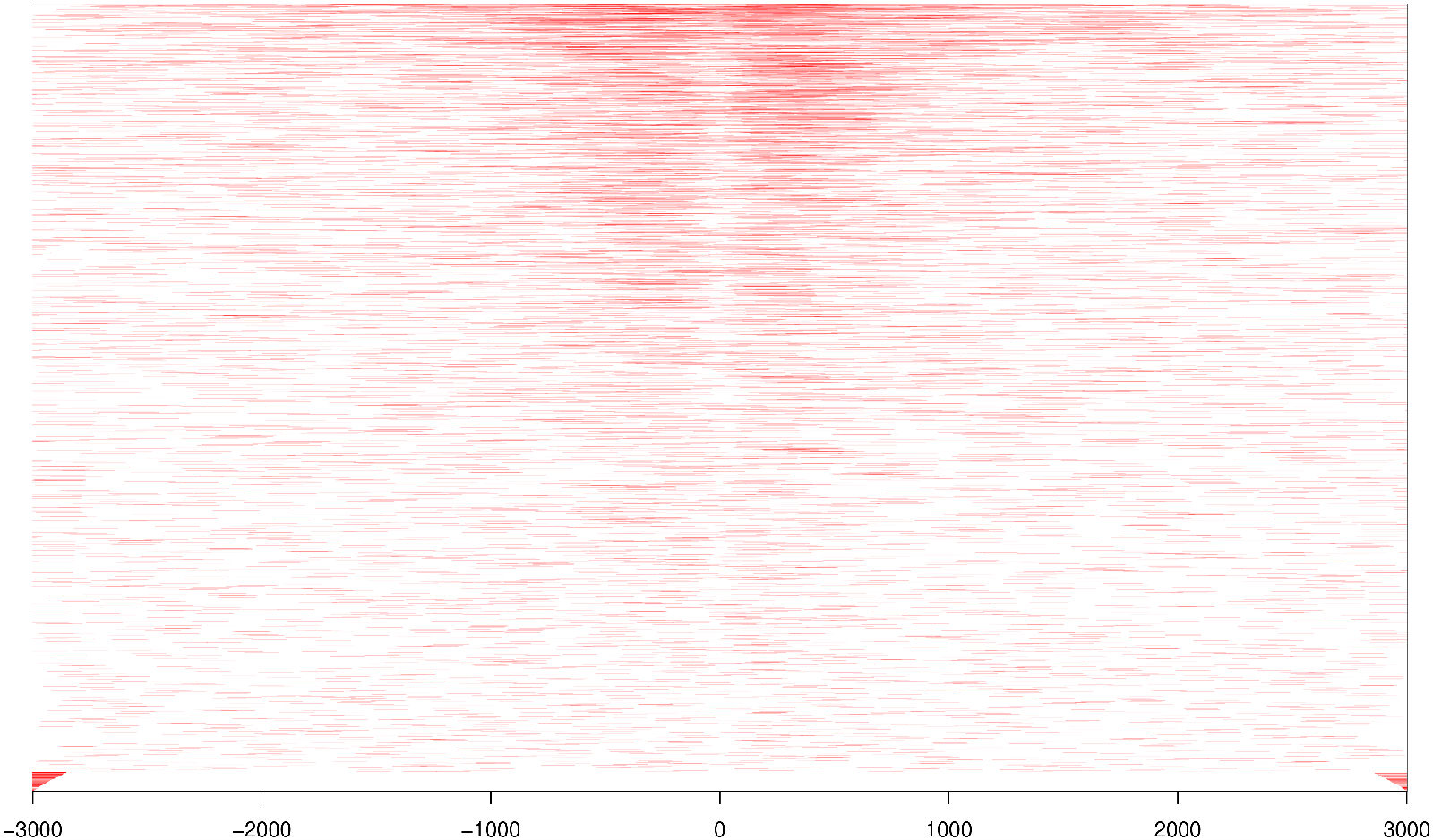
图 2 reads 在 TSS 近端富集强度的分布(热图分布)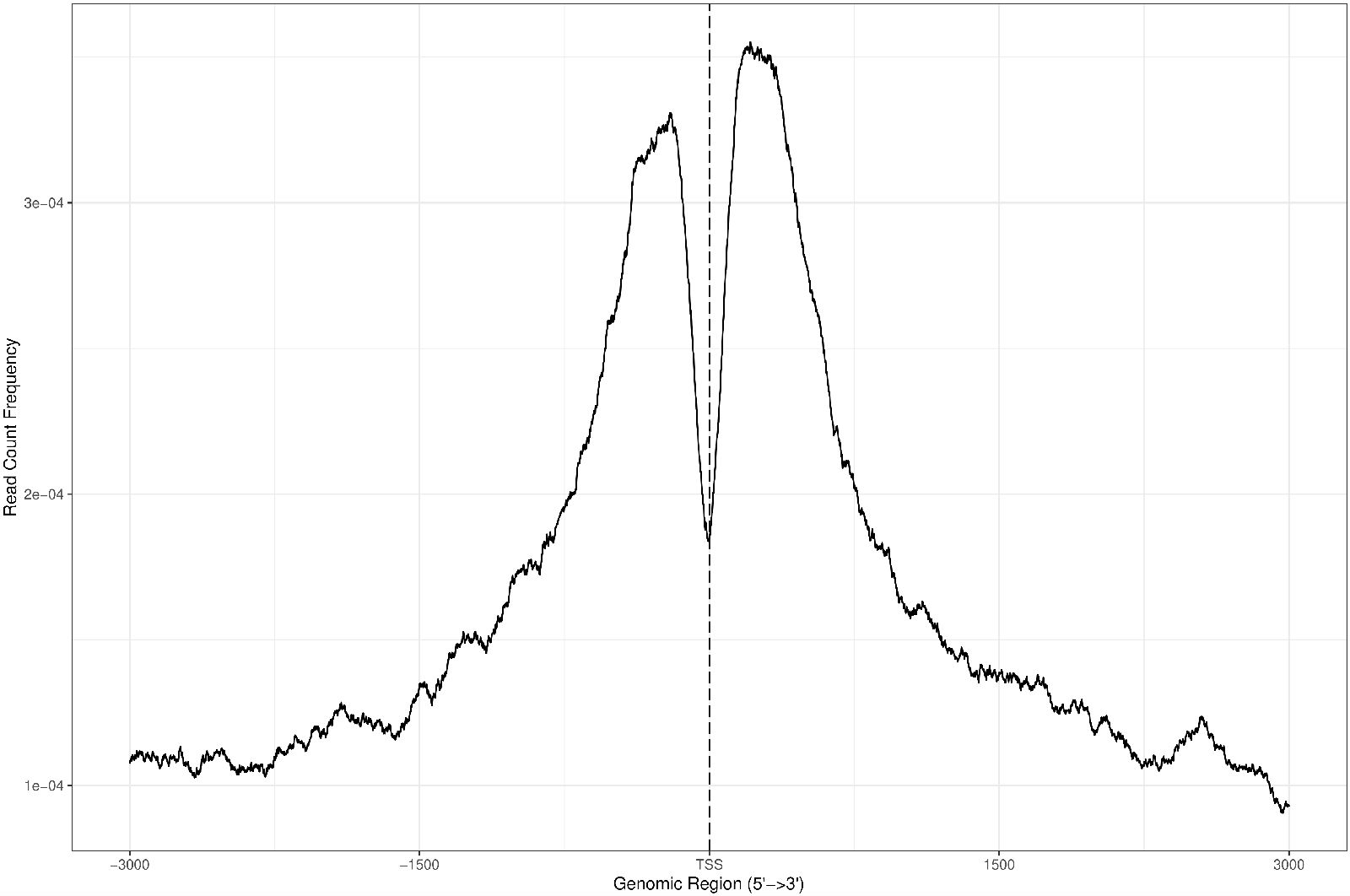
图 3 reads 在 TSS 近端富集强度的分布(峰图分布)
结果文件:
Results/2.5.tss_near/JFY.tagheatmap.pdf
Results/2.5.tss_near/JFY.plotavgprof.pdf
2.6. Reads 在 TSS 近端及远端富集强度分析
TSS 转录起始位点近端(0-3kb)及远端(10kb以上)的 reads 分布与特定的基因转录调控功能有关,统计 reads 在TSS 近端及远端的分布情况。
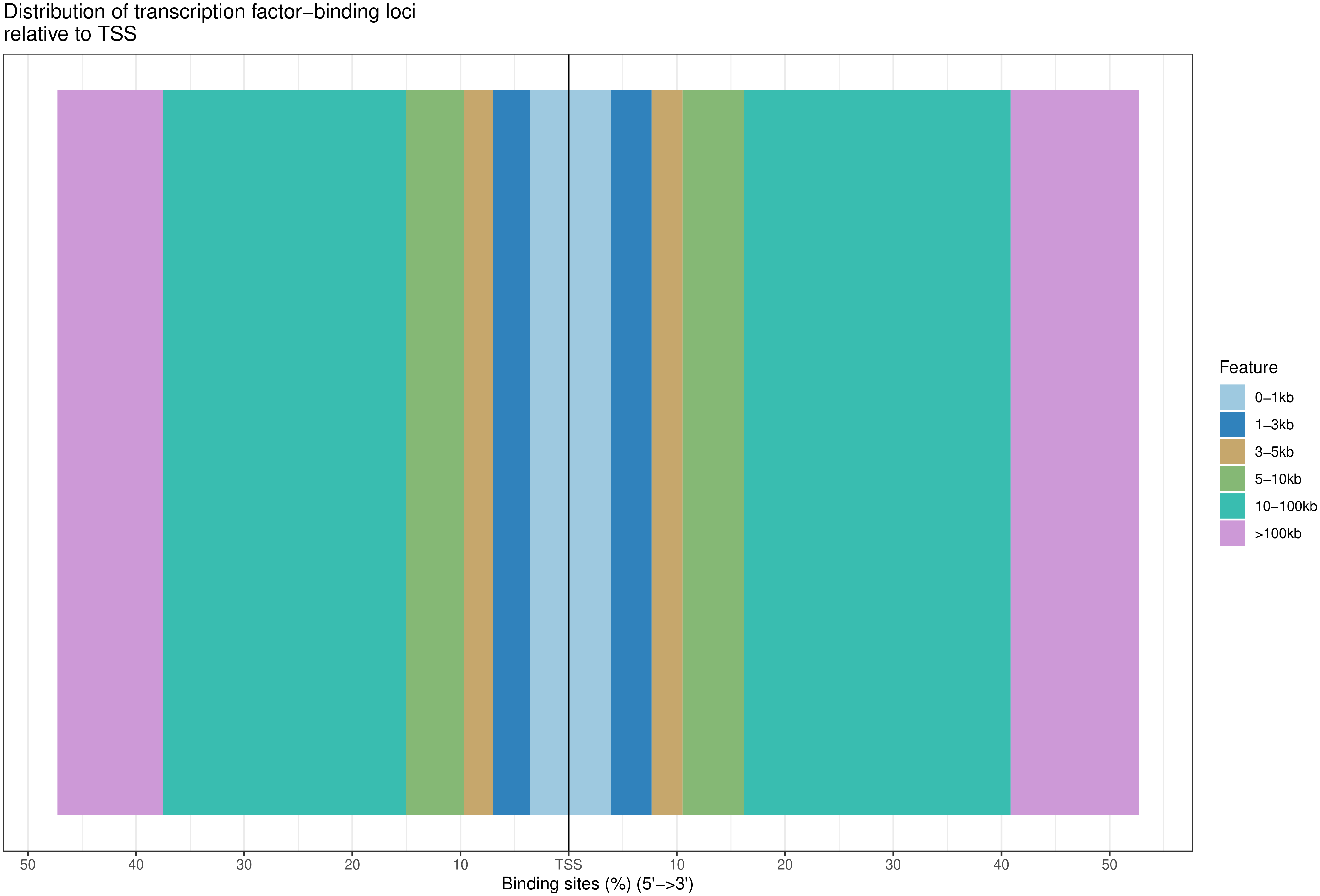
图4 reads 在 TSS 近端及远端富集强度的分布
结果文件:
2.7. Peak 在基因组上的分布
将 Peak 根据位置信息进行基因组注释基因结构元件,分别统计 Peak 在结构元件(intergenic region、upstream 5K、5`UTR、exon、intron,3’UTR、downstream5k)的数目,并根据其在各个元件上的富集程度,绘制分布特征。
Peak 在基因结构元件上的分布特征:

图5 Peak 在基因结构元件上的分布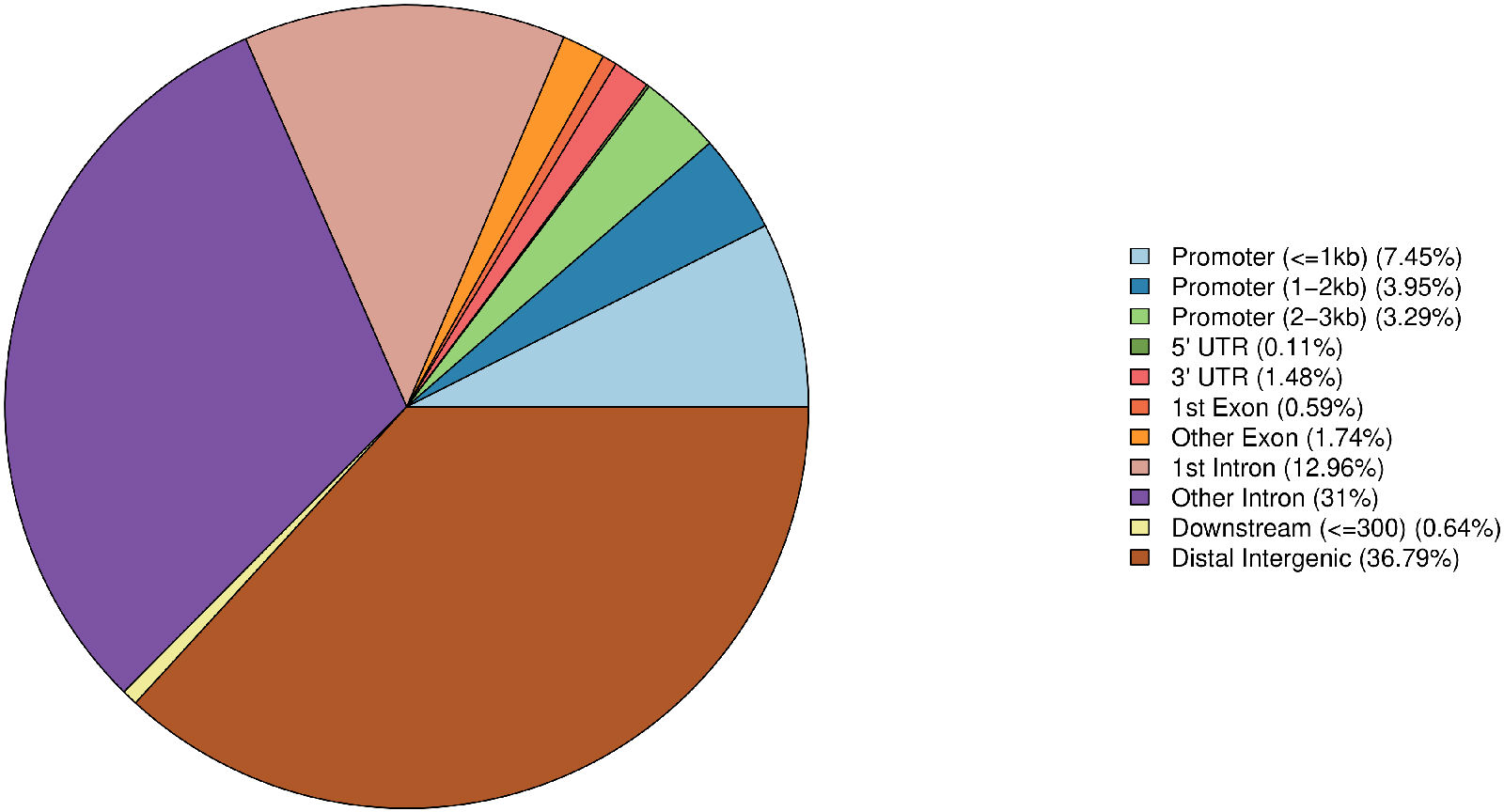
图6 Peak 在基因结构元件上的分布比例
Peak 在各基因结构元件上的交叉分布特征:
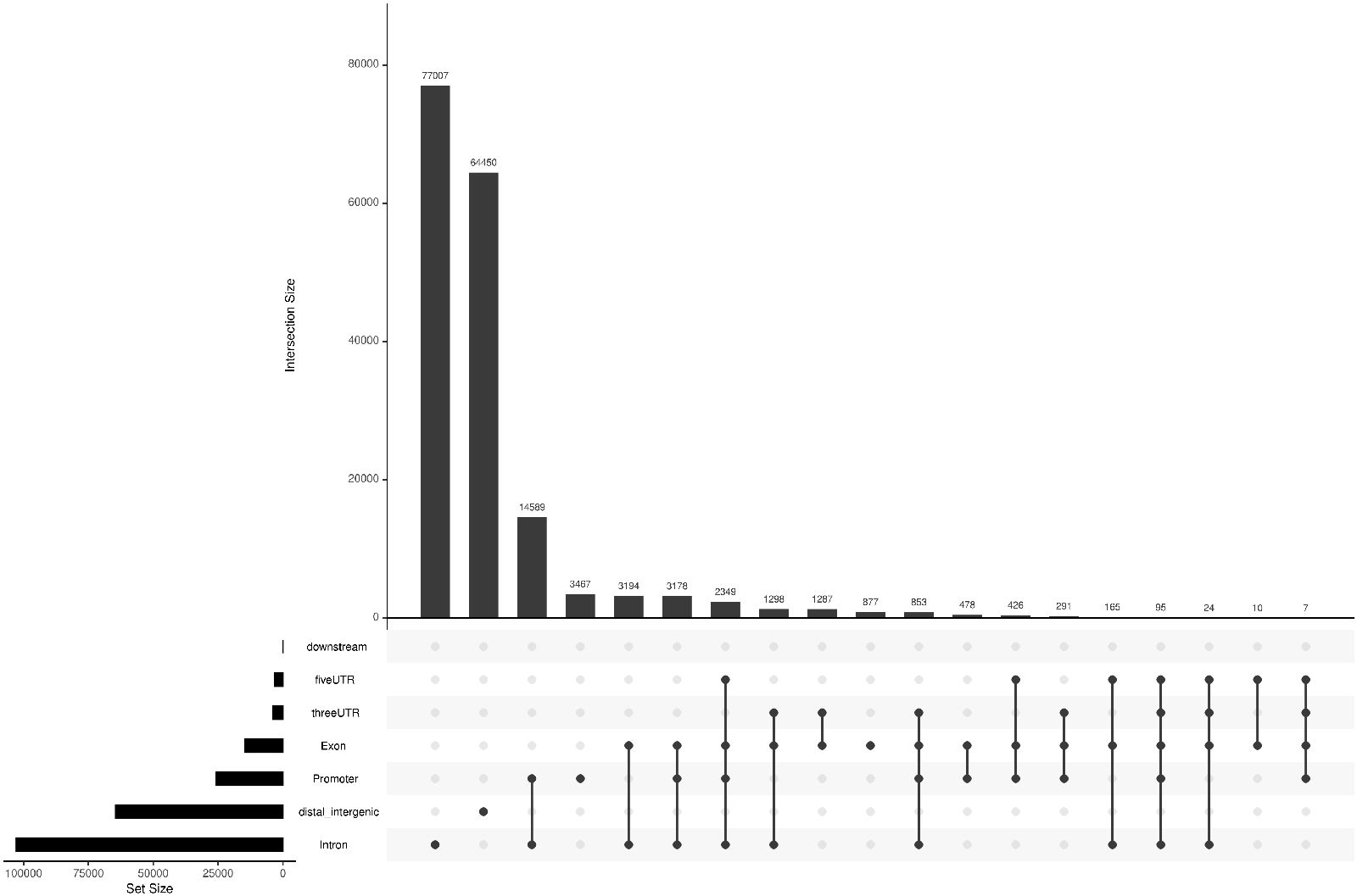
图7 Peak 在基因结构元件上的交叉分布(upsetplot)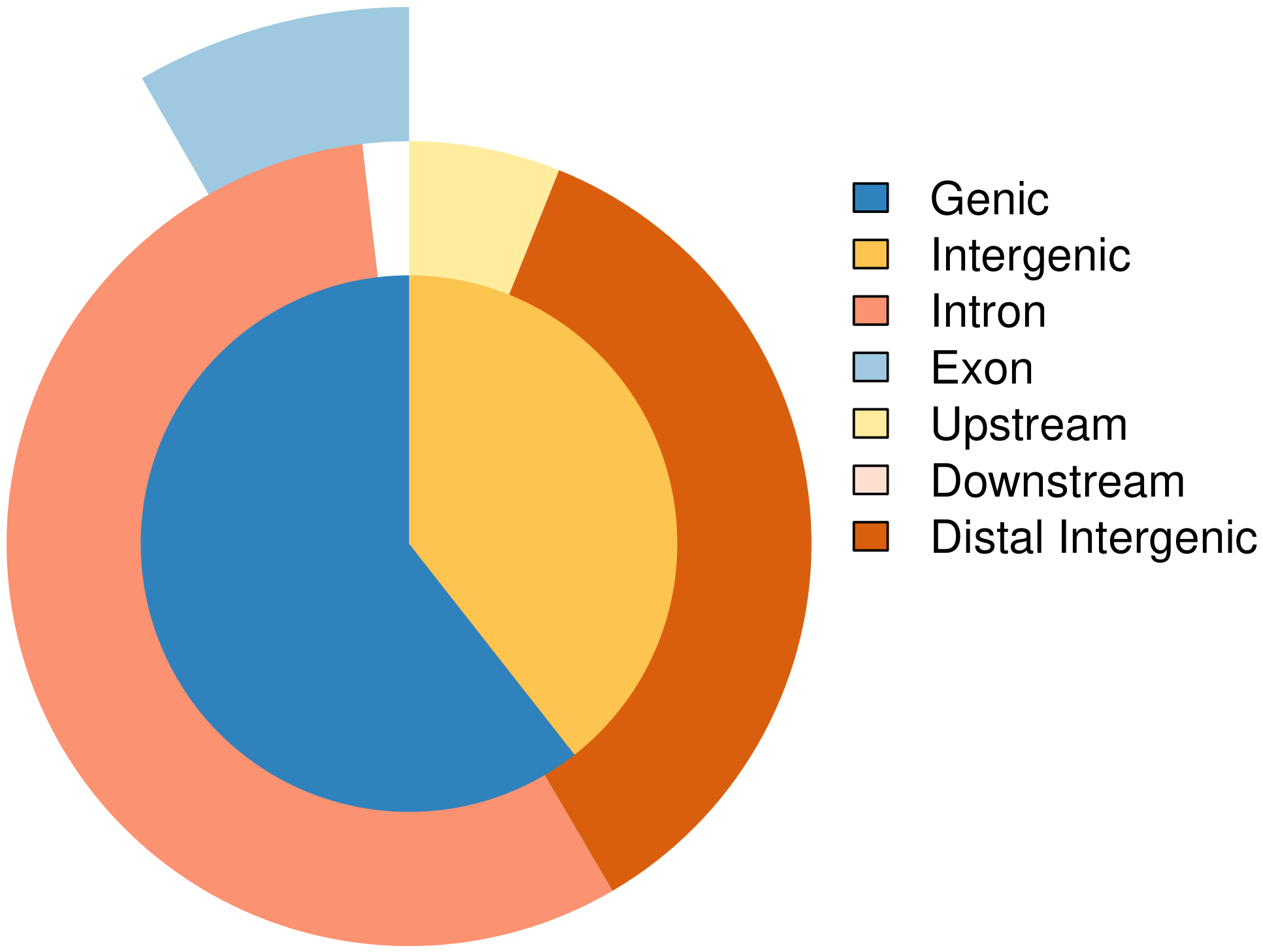
图8 Peak 在基因结构元件上的交叉分布(vennpie)
结果文件:
Results/2.7.peak_dis/JFY.peakAnnobar.pdf
Results/2.7.peak_dis/JFY.peakAnnopie.pdf
Results/2.7.peak_dis/JFY.peakAnnoupset.pdf
Results/2.7.peak_dis/JFY.peakAnnovinnpie.pdf
2.8. Peak 基因注释与 GO 功能分析
将 Peak 注释到基因promoter区,并对 Peak 相关基因进行GO 功能分析,并按照基因功能进行聚类分析。y轴为基因的功能聚类,x轴为基因count数,颜色为校正p值。
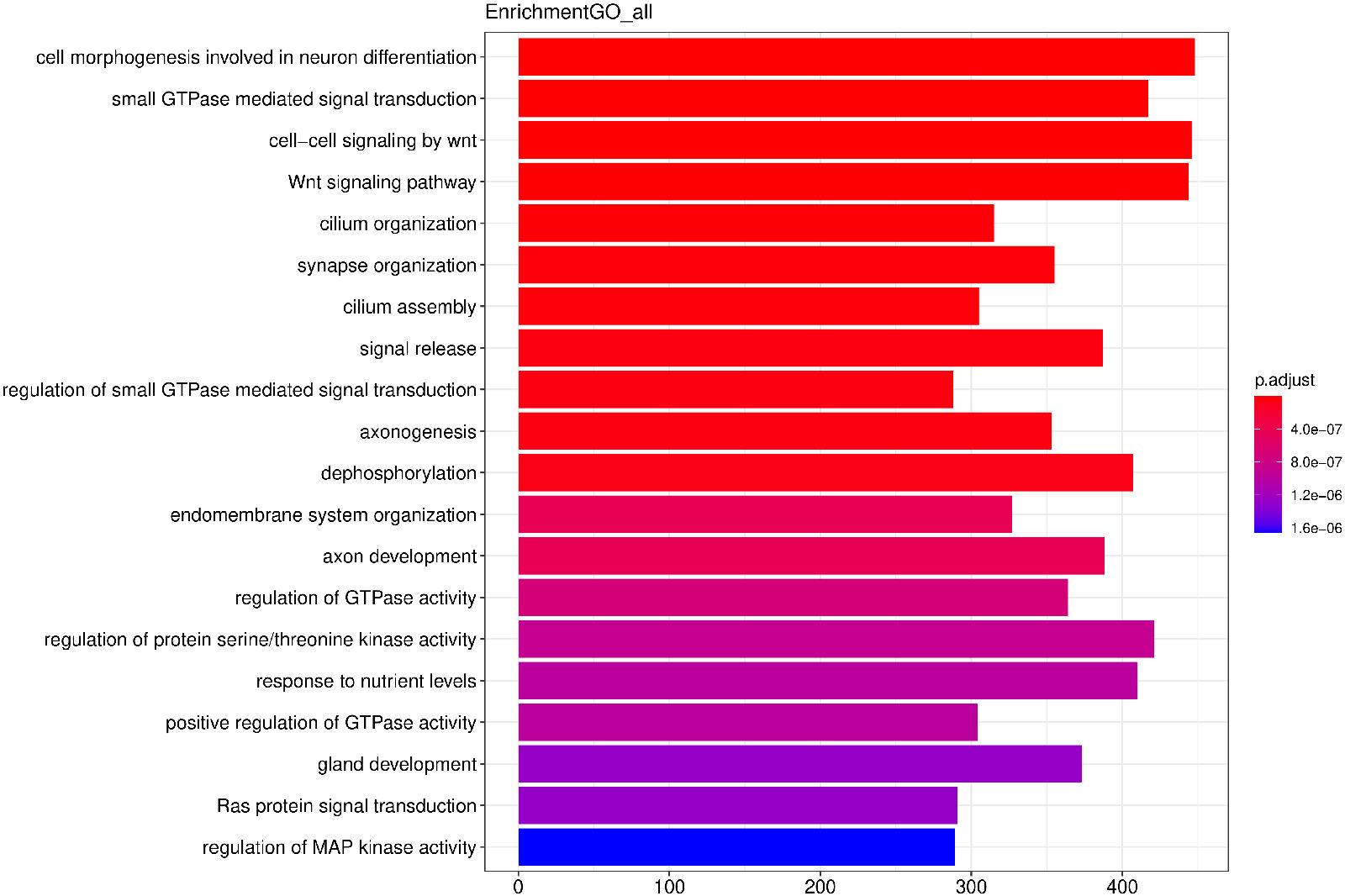
图9 Peak 相关基因的GO 功能富集分析(条形图)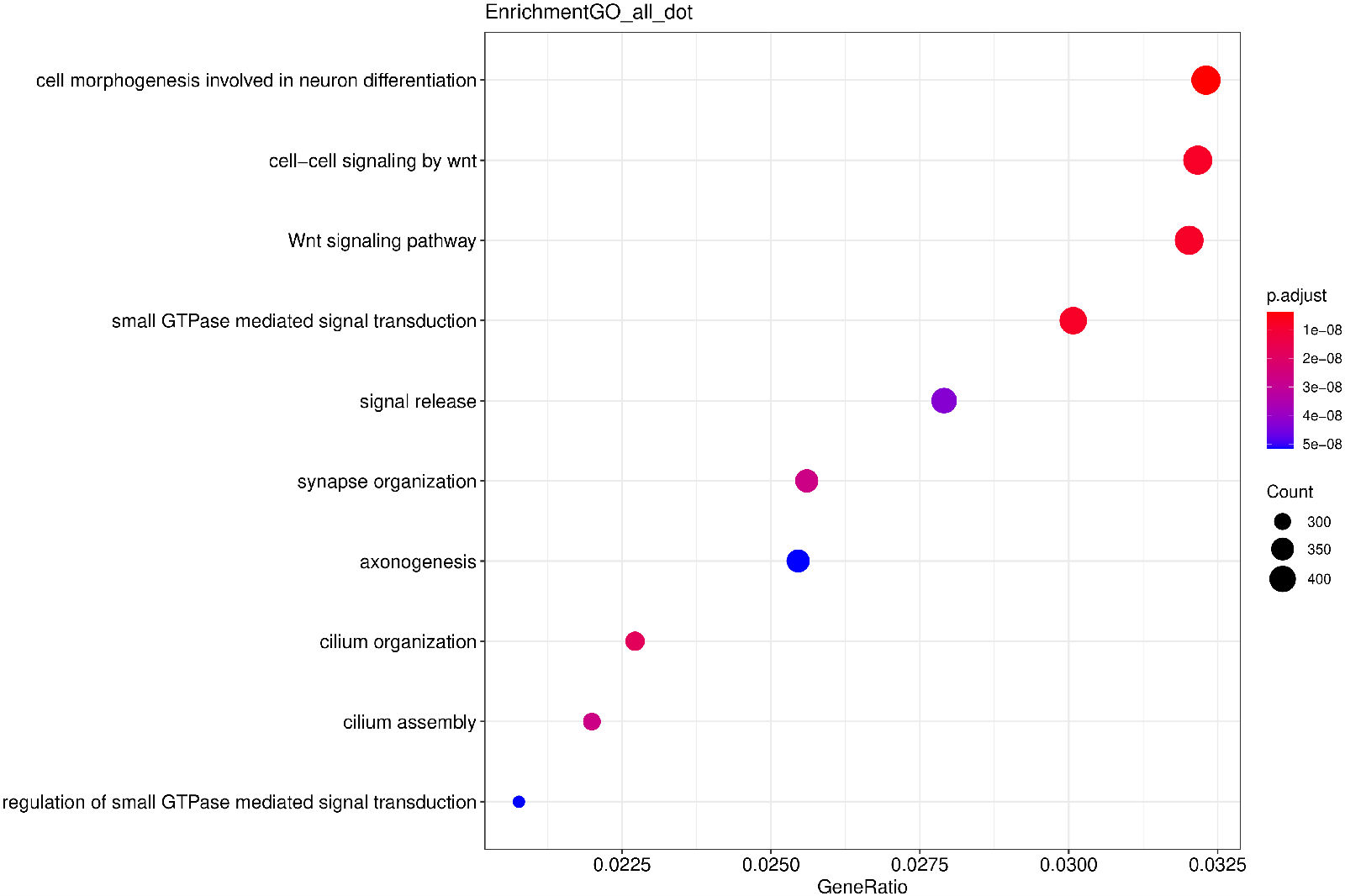
图10 Peak 相关基因的 GO 功能富集分析(气泡图)
结果文件:
Results/2.8.go/JFY.ego_ALL.csv
Results/2.8.go/JFY.GO-barplot.pdf
Results/2.8.go/JFY.GO-dotplot.pdf
2.9. Peak 基因注释与 KEGG 通路分析
将 Peak 注释到基因promoter,并对 Peak 相关基因进行GO 功能分析,并按照基因功能进行聚类分析。y轴为基因的功能聚类,气泡大小为基因count数,颜色为校正p值。
Pathway1中对peaks进行基因注释,仅采用临近基因注释。
Pathway2中对peaks进行基因注释,需要考虑多个因素,包括注释基因的外显子/内含子,promoter区,也包括peaks两侧可能包含顺式调控元件的区域。
Pathway1:
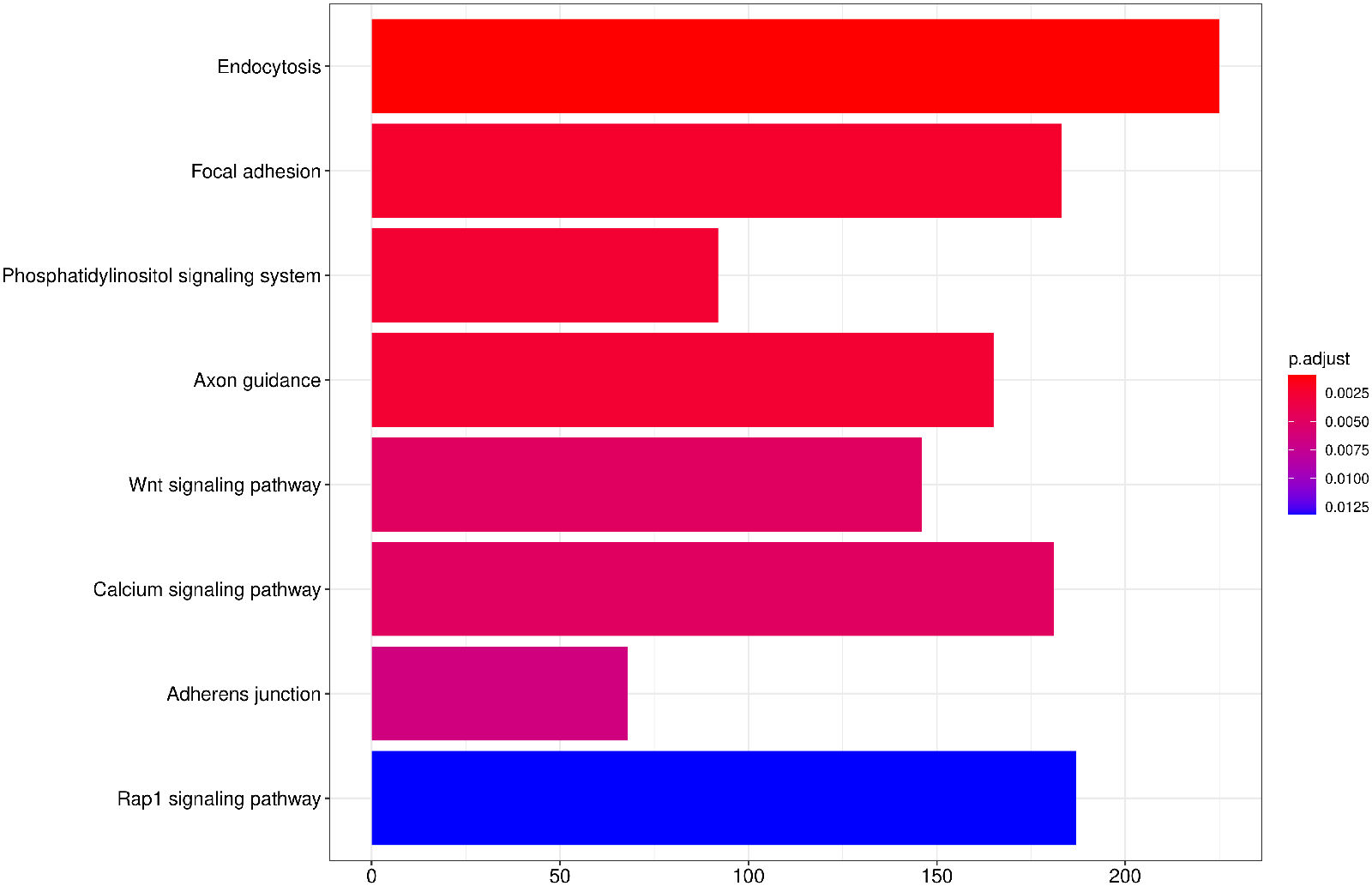
图11 Peak 相关基因的KEGG通路富集分析(条形图)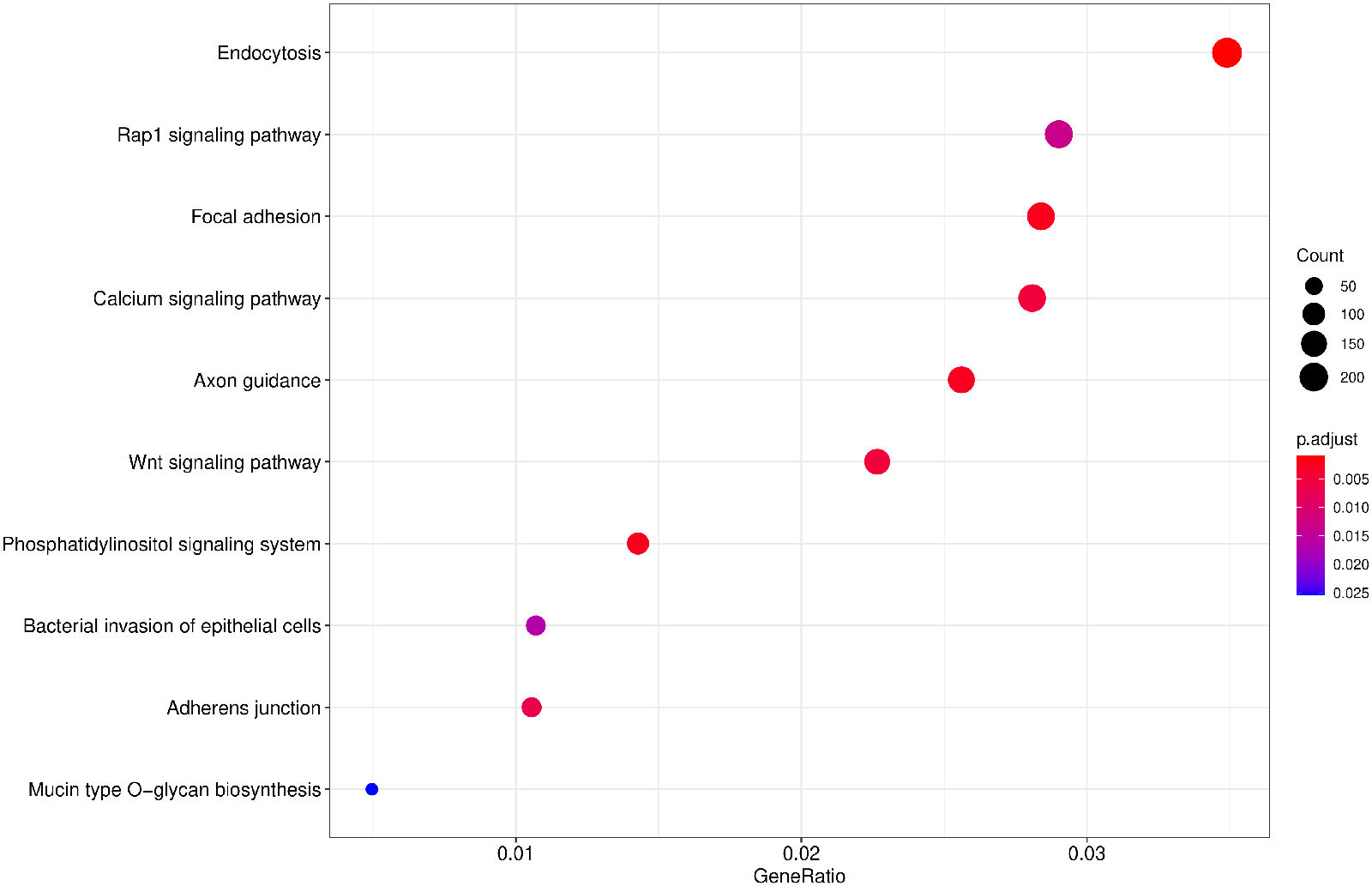
图12 Peak 相关基因的KEGG通路富集分析(气泡图)
Pathway2:
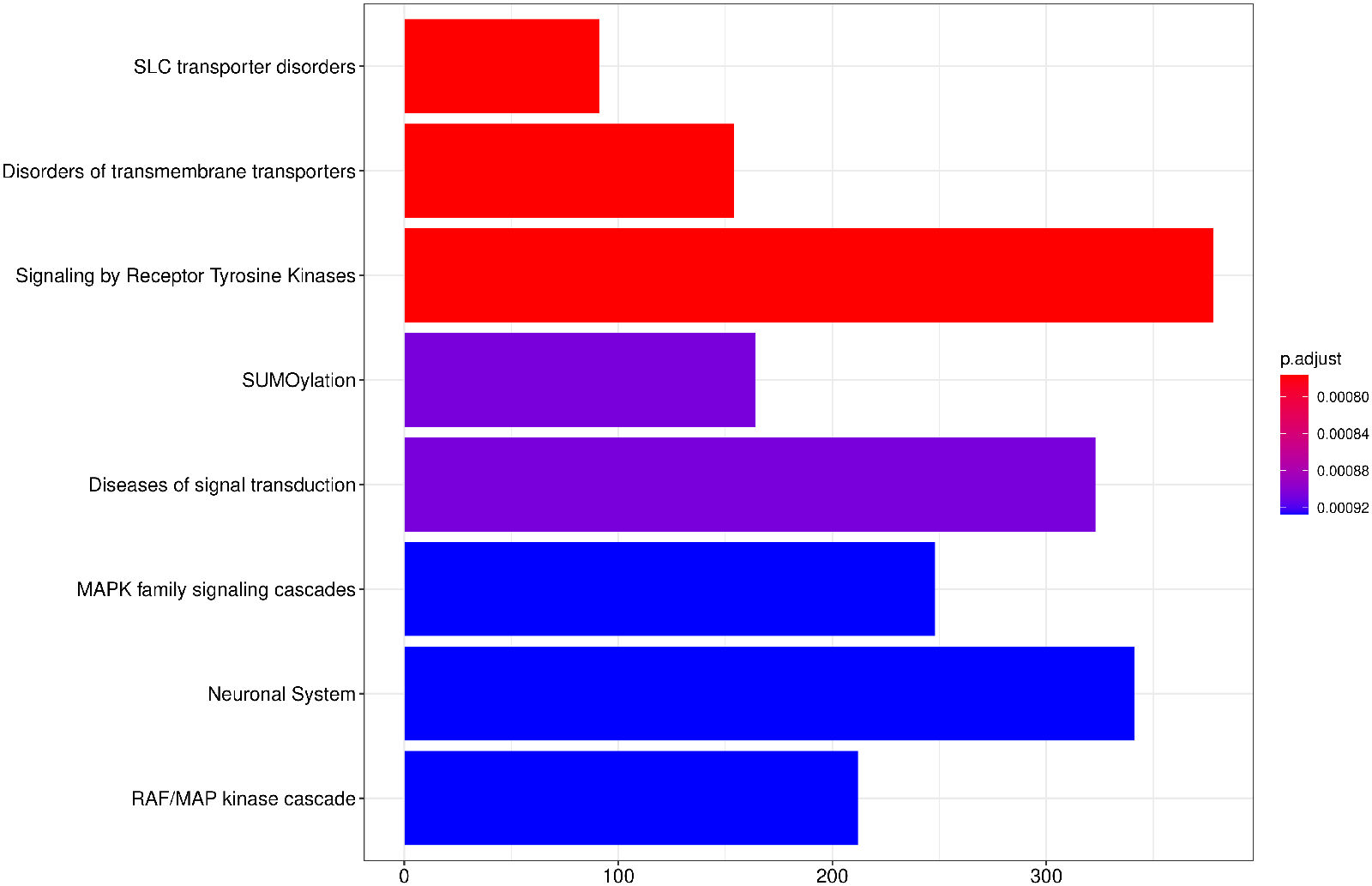
图13 Peak 相关基因的KEGG通路富集分析(条形图)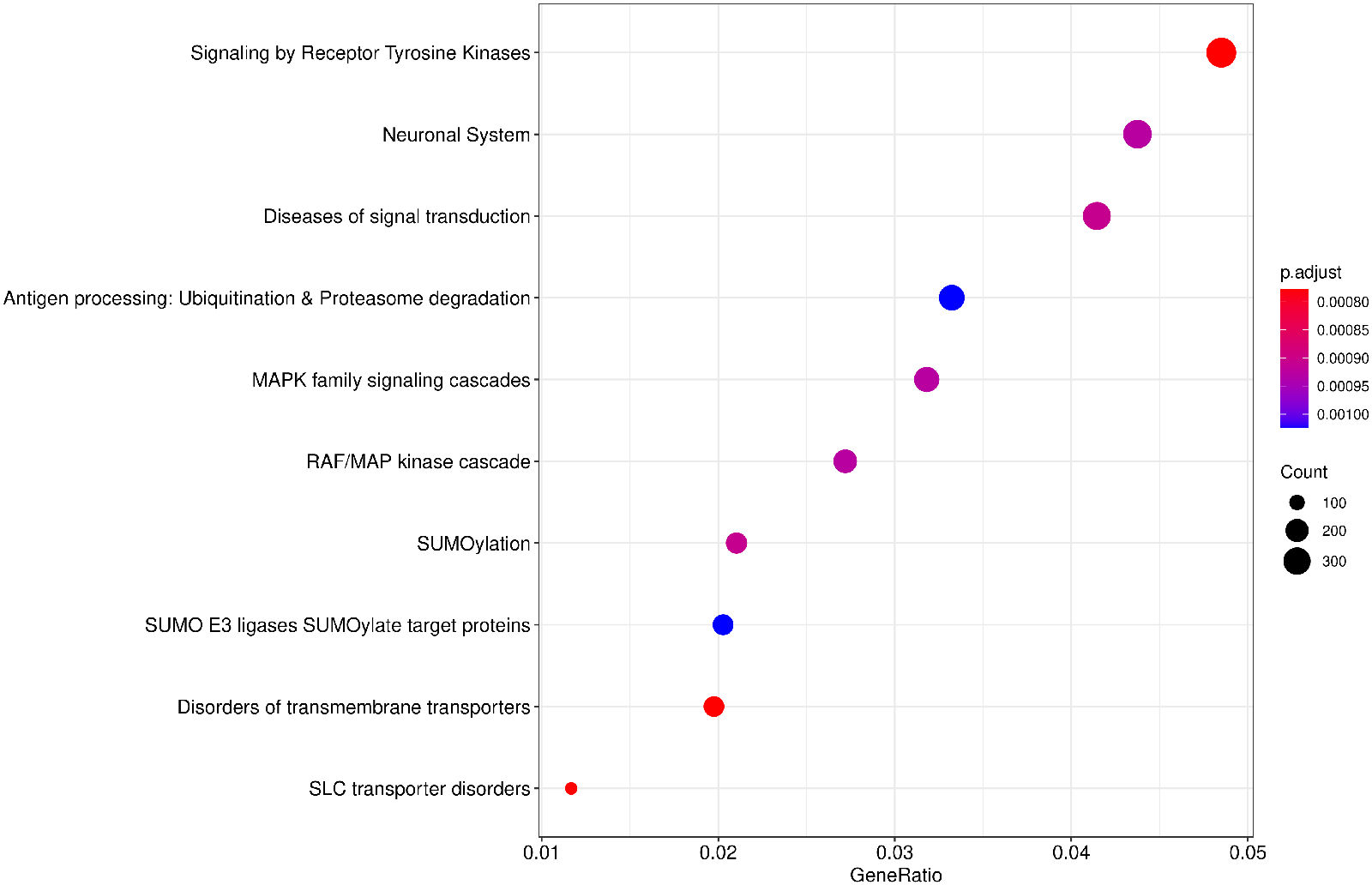
图14 Peak 相关基因的KEGG通路富集分析(气泡图)
结果文件:
Results/2.9.kegg/JFY.KEGG**.csv
Results/2.9.kegg/JFY.pathway**.pdf
2.10. Peak 区域 Motif 分析
用 Tomtom 软件对 Peak 区域鉴定 motif 序列;并将得到的 motif 序列与 JASPAR 数据库(JASPAR CORE 2016 database)进行比对,鉴定已知的 motif。
Tomtom 结果示例:
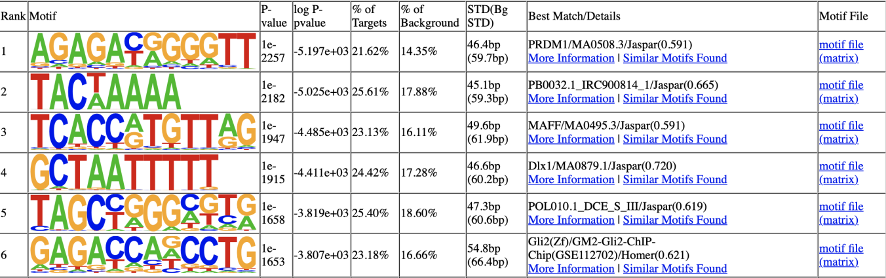
结果文件:
Results/2.10.motif/homerResults.html
Results/2.10.motif/knownResults.html
附 录
关于赛诚生物
广州赛诚生物科技有限公司是一家专注于科研基础实验服务及基因测序相关服务的生物技术公司。公司已有成熟的ChIP-seq,RNA-seq,甲基化及各种文库构建服务流程,具有丰富的文库构建和测序分析经验。同时,公司也开发多项前沿的实验技术,现已形成完善的ATAC-seq,CUT&TAG-seq体系。
除此之外,在基础科研服务方面,我们也有巨大优势。传统优势服务包括RNA pull down,DNA pull down,RIP,co-IP,细胞功能,流式检测,FISH等。最近刚刚兴起的ChIRP实验已成为我们的主流产品,他可以同时检测DNA、RNA、蛋白三者互作。
因此,我么拥有一支专注以“表观组学”为核心的医学转化技术服务团队,团队丰富的的科研经验涵盖了表观基因组学和基因组学中的各个方面,对多个组学的数据分析和挖掘有着丰富的 经验和全面的理解,比如:基因组修饰、染色质调控、基因表达调控、基因组变异等,我们 致力于提供领先的个性化的表观组学为核心的多组学整体解决方案。
联系我们
广州赛诚生物科技有限公司
地 址 :广州市黄埔区掬泉路3号B栋402房
电话:18664506006
网 站 : http://www.gzscbio.com
技术支持:
zhipeng.qian@gzscbio.com (137-6084-8131) 钱经理
